Biblioteca Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
Sanità Pubblica Veterinaria: Numero 72, Giugno 2012 [http://www.spvet.it/] ISSN 1592-1581
Documento reperibile all'indirizzo: http://spvet.it/indice-spv.html#554
Tecnologie diagnostiche innovative basate su biosensori applicate in campo per la diagnosi rapida di malattie - Innovative diagnostic biosensors based technologies applied in the field, for rapid diagnosis of diseases
Biagetti M., Orena M., Cuccioloni M.
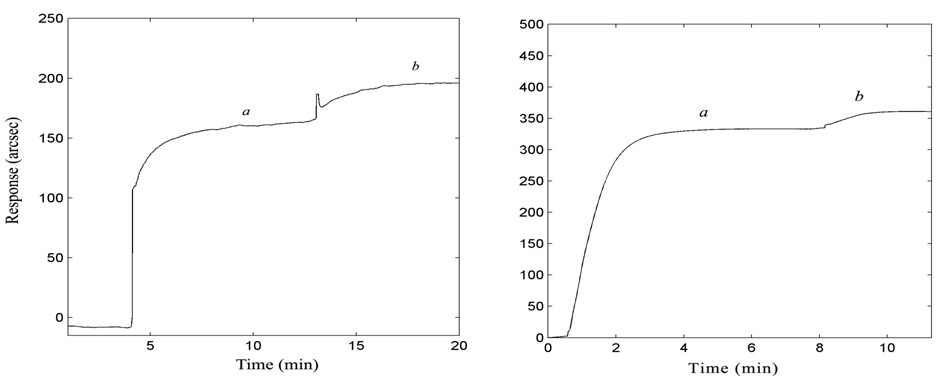
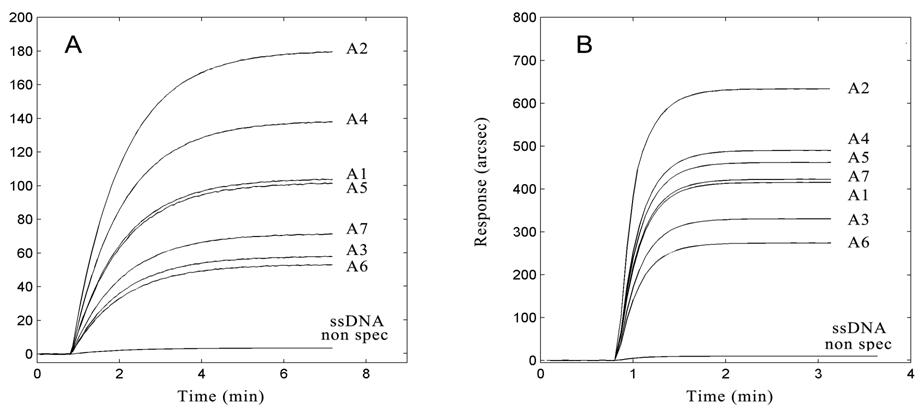
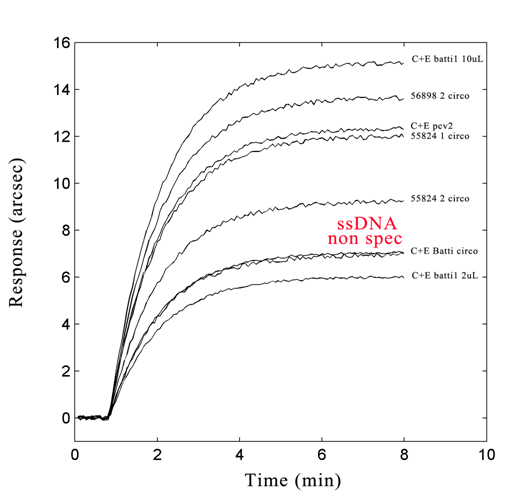
Abstract. The purpose of this paper is to determine an innovative way to perform diagnostic tests through the use of a field - biosensor. The device was connected to a laptop, in order to perform DNA analysis in a typical setting of a veterinary survey in the farm. This is possible thanks to a particular method of extraction, which allow the preparation of the sample in situ. This work, which is part of the research line: "innovative diagnostic technologies" of the Zooprofilattico UM Institute (Italy), shows the feasibility of applying this kind of biosensors for the diagnosis of viral diseases
Riassunto. Il presente lavoro è finalizzato a valutare l'efficacia di una modalità innovativa per l'esecuzione di accertamenti diagnostici tramite l'uso di un biosensore "da campo". Il sistema studiato è stato collegato ad un PC portatile e funziona in condizioni operative tipiche dei rilevamenti in azienda. Ciò è possibile grazie a una particolare metodica di estrazione che consente la preparazione del campione direttamente in sito. Il presente lavoro che si inserisce nella linea di ricerca "tecnologie diagnostiche innovative" dell'Istituto Zooprofilattico Sperimentale UM; mostra la fattibilità dell'applicazione di questo tipo di biosensori per la diagnosi di malattie virali