Sanità Pubblica Veterinaria: Numero 71, Aprile 2012 [http://www.spvet.it/] ISSN 1592-1581
Documento reperibile all'indirizzo: http://spvet.it/indice-spv.html#548
Validazione di un metodo analitico in Real Time PCR per la quantificazione di ingredienti di specie vegetali in matrici complesse: studio preliminare -
Validation of a Real Time PCR method for the quantification of vegetable ingredients in complex matrices like foods and feeds: a preliminary study.
Ciarrocchi F., Canonico C., Trozzi C., Gatto F., Briscolini S., Blasi G.
Download, file pdf [ Validazione di un metodo analitico in Real Time PCR 204 KB]
Abstract. The results of a preliminary validation of a Real Time PCR method for the quantification of soybean in complex vegetal matrices like foods and feeds are reported. The evaluation of the method was carried out, in terms of specificity, limit of detection (LOD), limit of quantification (LOQ) and repeatability. The research was developed in cooperation with three laboratories that received a detailed protocol for the reaction mixture preparation, the plate set up, samples and reagents.
The three laboratories performed successfully the assessment in order to estimate intermediate repeatability, LOD and LOQ. One of the two calibration curve was considered fit for the quantitation of the soybean ingredient
Riassunto. Sono presentati i risultati della validazione di un metodo analitico in Real Time PCR per la quantificazione, in campioni costituiti da più ingredienti, della percentuale di una singola specie vegetale presente, con particolare riferimento all'ingrediente soia. Lo studio ha coinvolto tre laboratori ed ha previsto la preparazione dei materiali di riferimento contenenti una definita combinazione di specie vegetali e percentuali scalari di soia no GM. Il DNA estratto da questi materiali è stato utilizzato per preparare le curve di calibrazione per quantificare la soia presente in un campione in analisi. Le curve di calibrazione (Curva A e Curva B) sono state saggiate da tre laboratori per verificare il LOD, LOQ e l'accuratezza in termini di ripetibilità.
Dai saggi effettuati è stato possibile determinare il LOD ed il LOQ del metodo, che sono risultati rispettivamente di 0,1 % e 1 %, mentre la migliore curva di calibrazione da impiegare nella quantificazione di una singola specie in matrici complesse è risultata la Curva A
Introduzione
Uno dei principali problemi riguardanti la coltivazione e commercializzazione di Organismi Geneticamente Modificati (OGM) nella filiera agroalimentare è rappresentato dalla possibile presenza di piccole tracce di OGM in mangimi formulati con materie non GM. La percentuale di tali contaminazioni, tecnicamente inevitabili, a volte supera la soglia di tolleranza per l'etichettatura stabilite dalla normativa comunitaria (Regolamento CE n. 1829/2003), determinando l'applicazione delle dovute sanzioni e denunce.
La normativa vigente, infatti, impone l'obbligo dell'etichettatura per percentuali di OGM autorizzati superiori allo 0.9 % rispetto ai singoli ingredienti di alimenti e mangimi. Tale obbligo non si applica a percentuali di OGM 0.9 %, purché la presenza sia accidentale o tecnicamente inevitabile. Gli operatori devono pertanto dimostrare di aver preso tutte le misure appropriate per evitarne la presenza.
Nel settore mangimistico esiste inoltre un fattore di tolleranza legato alle impurità botaniche, occasionalmente presenti come residui di lavorazioni precedenti o contaminazioni accidentali delle materie prime. La Direttiva 96/25/CE stabilisce tale soglia di tolleranza nell'ordine del 5% della materia prima e di conseguenza, tali contaminazioni non sono soggette ad etichettatura.
L'adozione di metodiche analitiche caratterizzate da un'elevata sensibilità, quali le analisi biomolecolari, ha portato sempre più frequentemente al rilevamento della presenza di tali impurità botaniche; talvolta queste contaminazioni possono essere costituite da OGM, creando un conflitto normativo tuttora irrisolto. L'analisi per il contenuto di OGM si è considerevolmente consolidata con l'introduzione dell'obbligatorietà di vincolare l'autorizzazione degli eventi di trasformazione alla disponibilità di materiali di riferimento e di metodi validati dal Laboratorio di Riferimento Europeo (http://gmo-crl.jrc.ec.europa.eu) (Reg. CE 1829/2003). Nonostante i numerosi progressi registrati negli ultimi anni, la valutazione del contenuto relativo delle specie vegetali, al fine di valutare se la presenza sia accidentale o meno, rappresenta tuttora un aspetto controverso, soprattutto nel caso in cui tale contaminazione è riconducibile a matrici OGM.
Sarebbe quindi auspicabile per i laboratori la definizione di strumenti analitici per la valutazione del rispetto dei limiti di tolleranza per le impurità botaniche, e quindi la stima del contenuto relativo delle varie specie vegetali che costituiscono il campione.
Alla luce di tali considerazioni, questo lavoro si è posto come obiettivo quello di ottenere uno strumento analitico che consentisse di avere dati che potessero avvicinarsi il più possibile a quella che è la reale situazione a livello produttivo.
E' stata pertanto validata una metodica Real Time PCR in grado di quantificare, in campioni costituiti da più ingredienti, la percentuale della singola specie vegetale presente, con particolare riferimento all'ingrediente soia. La ricerca ha previsto il coinvolgimento di tre laboratori ed ha permesso di verificare il LOD, LOQ e l'accuratezza del metodo in termini di ripetibilità.
Materiali e metodi
La scelta delle materie prime da impiegare in questa ricerca è stata eseguita sulla base dei risultati di un precedente lavoro in cui sono state saggiate diverse specie vegetali, impiegate nella formulazione di mangimi, per la verifica iniziale della purezza in termini di assenza di contaminazione da soia.
La preparazione dei materiali di riferimento è stata condotta mediante estrazione del DNA dalle materie prime mais, soia, girasole e crusca, secondo quanto riportato nella Norma ISO 21571:2005 - Annex A.3 e B.1-B.2. Dalla massa, completamente omogeneizzata mediante omogeneizzatore Osterizer 6805-50 (Steroglass, Perugia, Italia), sono stati allestiti due sottocampioni. Nel caso della soia non si è proceduto alla predisposizione dei sottocampioni. Da ciascun sottocampione e dal campione di soia sono stati prelevati 2 g di materiale per l'estrazione del DNA mediante trattamento chimico ed enzimatico (CTAB precipitante) e sua purificazione con kit commerciale (metodo CTAB/QIAGEN). Il DNA di tutti gli estratti è stato quantificato mediante analisi fotometrica e, ove necessario, diluito alla concentrazione di 30 ng/µL. Gli estratti così ottenuti sono stati riuniti in un'unica provetta, differente per ciascuna materia prima.
Gli stessi sono stati impiegati per l' allestimento, in sessioni indipendenti, di 2 curve di calibrazione (Curva A e Curva B), basate su 4 diversi punti. In totale sono stati predisposti 8 preparati, 4 per la Curva A e 4 per la Curva B, costituiti da DNA di soia rispettivamente al 50 %, 10 %, 5 %, 1 %, unito al DNA eterologo (diverso dal DNA di soia) uguale per tutte le 8 preparazioni e costituito da DNA di mais (50 %), crusca (33 %) e girasole (17 %). La specifica formulazione del DNA eterologo è stata scelta in base ai risultati di un precedente studio di ottimizzazione delle condizioni di amplificazione e di valutazione dell'influenza della quantità della singola materia prima all'interno della matrice complessa.
Prima di procedere alla preparazione dei materiali di riferimento, si è ritenuto opportuno condurre un ulteriore saggio in Real Time PCR Monitor Run per il gene lectina al fine di escludere una eventuale inibizione della reazione da parte di tale formulazione. La reazione è stata condotta allestendo una miscela di reazione di 25 µL contenente: TaqMan Universal Master Mix (Applied Biosystems, Foster City, CA) 1X; 0.9 µM di ciascun primer; DNA templato 5 µL. In Tabella 1 sono riportate le sequenze dei primer e della sonda impiegati, mentre il ciclo termico è stato impostato come segue: preattivazione UNG (Uracil-N-glicosilasi) a 50 °C per 2 minuti; preattivazione DNA polimerasi e denaturazione DNA a 95 °C, 10 minuti; 45 cicli di denaturazione a 95 °C per 15 secondi, annealing e sintesi a 60 °C per 60 secondi.
| Tabella 1 - Sequenze di primers e sonda impiegate per il saggio in Real time PCR per il sistema endogeno (lectina) | ||
|---|---|---|
| Nome | Tipo | Sequenza 5'-3' |
| GMax | F Fw | 5' - CTT TCT CGC ACC AAT TGA CA - 3' |
| GMaxR | Rv | 5' - TCA AAC TCA ACA GCG ACG AC - 3' |
| Probe GMax | Pr | 5'-Vic - CCA CAA ACA CAT GCA GGT TAT CTT GG - Tamra - 3' |
Tutti i preparati sono stati quindi formulati in modo da raggiungere una concentrazione finale di DNA di 7.5 ng/µL.
I materiali di riferimento così allestiti, insieme ai reagenti necessari per l'esecuzione delle analisi, sono stati inviati in regime di temperatura controllata ai laboratori coinvolti nello studio di validazione, e in particolare:
- Laboratorio 4 (LAB 4) per la verifica della ripetibilità e del LOD (Limite di determinazione), e del LOQ (Limite di quantificazione);
- LAB 3, LAB 4 e LAB 11 per la determinazione della curva di calibrazione migliore.
Ciascun laboratorio ha condotto la Real Time PCR per il sistema endogeno (lectina) impiegando i materiali di riferimento della Curva A e della Curva B, analizzando in triplicato ciascun punto delle curve, utilizzando i reagenti ed il ciclo termico sopra riportati.
Per la determinazione del LOD, eseguita dal solo LAB 4, è stata condotta la Real time PCR su diluizioni seriali di DNA di soia per ottenere 4 livelli di concentrazione (1 %, 0.1 %, 0.01 %, 0.005 %).
In particolare è stato impiegato il DNA di soia predisposto secondo le modalità sopra riportate e corrispondente al punto a più bassa concentrazione di soia (1 %). Da questo sono state allestite, in duplice copia, ciascuna per le due curve A e B, tre diluizioni scalari di DNA di soia, in DNA eterologo secondo le quantità riportate in Tabella 2.
| Tabella 2 - Materiali di riferimento allestiti per la determinazione del LOD e del LOQ | ||
|---|---|---|
| Materiali | Soia | DNA Eterologo |
| Punto 0.1% Soia | 2,5 µL (Soia 1%) | 22,5 µL |
| Punto 0.01% Soia | 2,5 µL (Soia 0,1%) | 22,5 µL |
| Punto 0.005% Soia | 1,25 µL (Soia 0,1%) | 23,75 µL |
Ogni diluizione è stata esaminata in Real time PCR in triplicato insieme ai 4 punti delle due curve di calibrazione, al fine di verificare la ripetibilità del metodo. Il calcolo del LOD è stato ricavato determinando il livello minimo di concentrazione del DNA di soia per cui almeno il 95 % dei replicati è risultato positivo.
Per la determinazione del LOQ sono stati utilizzati i livelli di concentrazione per i quali si sono ottenuti dei valori di Ct utili nel 100% dei replicati: 1%, 0,1%, 0,01%, 0,005%.
Il calcolo della % di soia presente in ognuno di questi campioni è stato eseguito attraverso la funzione di tendenza in cui il valore di Ct associato ad ogni punto della curva di calibrazione viene interpolato con il valore logaritmico della sua % così da ottenere la retta di interpolazione lineare.
Il valore di Ct di ogni campione incognito è stato inserito nell'equazione della retta in modo da essere calcolato il corrispondente valore %.
Per ogni replicato di ogni livello di concentrazione analizzato è stata calcolata la % di soia e per ogni livello di concentrazione è stato calcolato il valore medio (xm), lo scarto tipo di ripetibilità (sr) e la deviazione standard di ripetibilità relativa (RSDr). Il LOQ è definito come la più bassa concentrazione alla quale il valore della RSDr è ≤ 25%.
La ripetibilità è stata calcolata come deviazione standard di ripetibilità relativa (RSDr) per i 4 punti della curva di calibrazione (DNA di soia al 50 %, 10 %, 5 %, 1 %) utilizzando i risultati ottenuti dal LAB 4 con le curve di calibrazione A e B. Per ogni replica di tutti i punti di entrambe le curve è stata calcolata la % di soia utilizzando la retta di calibrazione costruita sulla base dei punti della curva a cui ogni replica non apparteneva. In questo modo per ogni punto della curva sono stati utilizzati 12 valori con i quali per ogni punto della curva sono stati calcolati la media (xm), lo scarto tipo di ripetibilità (sr), la deviazione standard di ripetibilità relativa (RSDr) ed il limite di ripetibilità (r = t * sr*√ 2; considerando 11 gradi di libertà il valore di t di Student è stato considerato pari a 2,20). Il criterio di accettabilità per la ripetibilità prevede che la RSDr sia ≤ 25% per tutti i punti della curva di calibrazione.
In accordo con quanto stabilito da ENGL (Regolamento della Commissione Europea N. 1829/2003 sui criteri di accettabilità delle analisi sugli OGM), per la scelta della migliore curva di calibrazione da impiegare nella quantificazione di un singolo ingrediente, le rette costruite da ciascun laboratorio sono state valutate in base ai seguenti parametri:
- linearità della reazione calcolando il valore medio del coefficiente di correlazione R2 delle rette di calibrazione ottenuto da tre prove indipendenti, una per ogni laboratorio coinvolto. Tale valore è ritenuto accettabile quando risulta maggior o uguale a 0.98;
- efficienza dell' amplificazione sfruttando la seguente relazione funzionale:
E (%) = [(10 -1/slope) -1] * 100
dove la pendenza (slope) della curva di calibrazione è la media dei valori di pendenza delle rette di calibrazione ottenuti da tre prove indipendenti. Il criterio di accettabilità è rispettato quando -3.1 ≥ slope ≥ -3.6.
Risultati e discussione
Nella Tabella 3 sono riportati i risultati della PCR lectina Monitor Run effettuata sulla formulazione del DNA eterologo, preparato in ragione del 50 % di mais, 33 % di crusca e 17 % di girasole ed utilizzato nell'allestimento dei punti delle curve di calibrazione.
| Tabella 3 - Real Time PCR (Th 0,24) | ||
|---|---|---|
| Formulazione DNA eterologo | Ct ± (Dev.st.) | ΔCt |
| DNA Tq (30 ng/ µL) | 27,00 ± (0,027) | 0,07 |
| DNA Tq (30 ng/ µL) | 26,96 ± (0,027) | 0,07 |
| DNA 1:4 (7,5 ng/ µL) | 27,97 ± (0,021) | 0,07 |
| DNA 1:4 (7,5 ng/ µL) | 27,94 ± (0,021) | 0,07 |
Malgrado il ΔCt notevolmente basso, la qualità delle curve relative alla concentrazione più bassa è stata considerata sufficientemente buona e si è ritenuto opportuno utilizzare tale concentrazione di DNA (7,5 ng/ µL) per le successive determinazioni. Nella Tabella 4 sono riportati i risultati per la determinazione del LOD eseguita dal LAB 4 su diluizioni seriali di DNA di soia per i 4 livelli di concentrazione (1 %, 0.1 %, 0.01 %, 0.005 %). Il LOD è risultato pari a 0,1 %.
| Tabella 4 - Risultati della determinazione del LOD eseguita dal LAB 4 | ||
|---|---|---|
| Diluizioni | Ottenuti/Attesi | % Rilevati |
| 1% | 6/6 | 100 |
| 0,10% | 6/6 | 100 |
| 0,01% | 5/6 | 83 |
| 0,005% | 2/6 | 33 |
Nella Tabella 5 sono riportati i dati per la determinazione del LOQ eseguita dal LAB 4 sui livelli di concentrazione 1 %, 0,1 %, 0,01 %, 0,005 %. Il LOQ è risultato pari a 1%.
| Tabella 5 - Risultati della determinazione del LOQ eseguita dal LAB 4 | |||||
|---|---|---|---|---|---|
| Diluizioni | Curva % | Xm | Sr | RSDr | |
| A | B | ||||
| 1% | 1,03 | 0,96 | 1 | 0,1 | 7,9 | 1,09 | 0,96 | 0,89 | 0,9 |
| 0,1% | 0,16 | 0,09 | 0,1 | 0 | 30,42 | 0,11 | 0,12 | 0,12 | 0,06 |
| 0,01% | 0,008 | 0,01 | 0 | 0 | 59,72 | - | 0,03 | 0,02 | 0,01 |
| 0,01% | 0,008 | 0,02 | 0 | 0 | 60,61 |
Nella Tabella 6 sono riportati i dati per la valutazione della ripetibilità eseguita dal LAB 4 utilizzando i 4 punti delle curve di calibrazione A e B (DNA di soia al 50 %, 10 %, 5 %, 1 %). I risultati per la ripetibilità per ogni punto della curva di calibrazione sono stati: Soia 1 % r = 0.4; Soia 5 % r = 1,2; Soia 10 % r = 2,5; Soia 50 % r = 7,5.
| Tabella 6 - Risultati della ripetibilità del LOQ eseguita dal LAB 4 | ||||
|---|---|---|---|---|
| % Soia | 1 | 5 | 10 | 50 |
| Curva A (I seduta) | 1 | 5,8 | 11 | 52 |
| 0,7 | 6,1 | 11 | 50 | |
| 1,1 | 5,7 | 10 | 49 | |
| Curva A (II seduta) | 1 | 5,3 | 11 | 47 |
| 1,1 | 5,5 | 11 | 46 | |
| 0,9 | 5 | 9,9 | 47 | |
| Curva B (I seduta) | 0,8 | 5,1 | 9,2 | 45 |
| 1 | 5,2 | 9,8 | 48 | |
| 1,1 | 5,5 | 8,6 | 44 | |
| Curva B (II seduta) | 1 | 5,2 | 10 | 51 |
| 1 | 4,8 | 9,9 | 49 | |
| 0,9 | 5,3 | 9,7 | 51 | |
| media | 1 | 5,4 | 10 | 48 |
| Sr | 0,1 | 0,4 | 0,8 | 2,4 |
| RSDr | 13 | 7 | 8 | 5 |
| r | 0,4 | 1,2 | 2,5 | 7,5 |
| r medio | 2,9 |
|||
sr=scarto tipo di ripetibilità; |
||||
Come confermato da recenti ricerche (Kodama et al., 2010) la ripetibilità dei metodi biochimici di quantificazione degli OGM, basati sulla Real Time PCR, è legata alla concentrazione dell'analita. A riprova di ciò, è stata eseguita una correlazione lineare tra le concentrazioni di soia (%) impiegate dal LAB 4 per la costruzione delle curve di calibrazione A e B, ed i relativi valori del limite di ripetibilità r (Figura 1). I risultati ottenuti hanno avvalorato l'ipotesi dell'esistenza di tale relazione con una correlazione del 68%.
Figura 1. Scatterplot e relativa retta di correlazione lineare
tra le concentrazioni di soia saggiate nella costruzione di entrambe
le curve di calibrazione ed i relativi valori del limite di ripetibilità r.
Nella Tabella 7 vengono riportati i risultati dei valori di Ct e relativa elaborazione statistica effettuata per ciascun punto delle due curve di calibrazione ottenute da ogni laboratorio coinvolto in questo lavoro.
| Tabella 7 - Risultati dei saggi in Real Time PCR condotti dai laboratori 3, 4 ed 11 per la validazione delle curve di calibrazione. | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Lab | Curva (Ct) |
Th | Negativo | Positivo Media ± (ds) |
||||||||||||||
Campione A |
Campione B |
|||||||||||||||||
| % soia | Log 10 [%] | Valore 1 | Valore 2 | Valore 3 | Media | DS | % soia | Log 10 [%] | Valore 1 | Valore 2 | Valore 3 | Media | DS | |||||
| 4 | 1 | 0,00 | 32,84 | und. | 32,74 | 32,79 | 0,07 | 1 | 0,00 | 33.25 | 32.89 | 32,73 | 32.96 | 0,27 | 0.02 | und. | 34.31 ± 0.08 | |
| 5 | 0,70 | 30,09 | 30,00 | 30,13 | 30,07 | 0,07 | 5 | 0,70 | 30,25 | 30,22 | 30,10 | 30,19 | 0,08 | - | - | - | ||
| 10 | 1,00 | 29,10 | 29,01 | 29,17 | 29,09 | 0,08 | 10 | 1,00 | 29,28 | 29,18 | 29,38 | 29,28 | 0,10 | - | - | - | ||
| 50 | 1,70 | 26,57 | 26,65 | 26,68 | 26,63 | 0,06 | 50 | 1,70 | 26,68 | 26,58 | 26,71 | 26,66 | 0,07 | - | - | - | ||
| 11 | 1 | 0,00 | 31,29 | 31,19 | 31,06 | 31,18 | 0,12 | 1 | 0,00 | 31,36 | 31,74 | 31,87 | 31,66 | 0,27 | 0,2 | und. | 32,47 ± 0,22 | |
| 5 | 0,70 | 29,37 | 29,43 | 29,50 | 29,43 | 0,07 | 5 | 0,70 | 28,76 | und. | 28,98 | 29,87 | 0,16 | - | - | - | ||
| 10 | 1,00 | 27,81 | 28,00 | 27,56 | 27,79 | 0,22 | 10 | 1,00 | 28,41 | 28,08 | 27,79 | 28,09 | 0,31 | - | - | - | ||
| 50 | 1,70 | 25,73 | 26,00 | 25,72 | 25,82 | 0,16 | 50 | 1,70 | 25,60 | 25,57 | 25,55 | 25,57 | 0,03 | - | - | - | ||
| 3 | 1 | 0,00 | 32,57 | 32,58 | 32,73 | 32,63 | 0,09 | 1 | 0,00 | 33,28 | 32,95 | 33,01 | 33,08 | 0,18 | 0,2 | und. | 39,16 ± 0,80 | |
| 5 | 0,70 | 30,21 | 30,24 | 30,15 | 30,20 | 0,05 | 5 | 0,70 | 30,20 | 30,15 | und. | 30,18 | 0,04 | - | - | - | ||
| 10 | 1,00 | 29,32 | 29,29 | 29,21 | 29,27 | 0,06 | 10 | 1,00 | 29,03 | 28,98 | 29,08 | 29,03 | 0,05 | - | - | - | ||
| 50 | 1,70 | 26,91 | 26,88 | 26,85 | 26,88 | 0,03 | 50 | 1,70 | 26,68 | 26,84 | 26,66 | 26,73 | 0,10 | - | - | - | ||
| und.= indeterminato | ||||||||||||||||||
Per ciascun punto della curva (1, 5, 10, 50% Soia) vengono riportati i valori di Ct raggiunti Dai risultati ottenuti dalle curve di calibrazione disegnate da ciascun laboratorio, è stato possibile osservare che tutte le curve hanno raggiunto un valore accettabile del coefficiente di correlazione R2, con valori sempre superiori o uguali a 0.98. Per ciascuna curva di calibrazione A e B il valore medio dei coefficienti R2 è rientrato nei criteri di accettabilità, raggiungendo rispettivamente i valori di 0.99 e 1.00.
L'efficienza di amplificazione è stata diversa in base alla curva. In particolare, mentre tutti i valori dello slope della curva A hanno raggiunto valori accettabili compresi tra -3.2 e -3.6, dimostrando un' efficienza di amplificazione tra l' 89 % ed il 104 %, quelli della curva B sono risultati inferiori e compresi tra -3.8 e -3.6. Tra questi ultimi, inoltre, è stato possibile osservare che solo per la curva B del LAB 11 è stato soddisfatto il criterio di accettabilità, contrariamente a quanto evidenziato nei valori dei LAB 3 e 4 per le quali lo slope è risultato maggiore di -3.6 e di conseguenza inaccettabile. I valori medi di slope delle curve di calibrazione A e B sono stati rispettivamente di -3.4 e -3.7, indicando una migliore efficienza di amplificazione nella curva di calibrazione A (97 %) rispetto alla curva B (87 %). Nella Figura 2 vengono mostrate le migliori rette di calibrazione ottenute in questa fase.
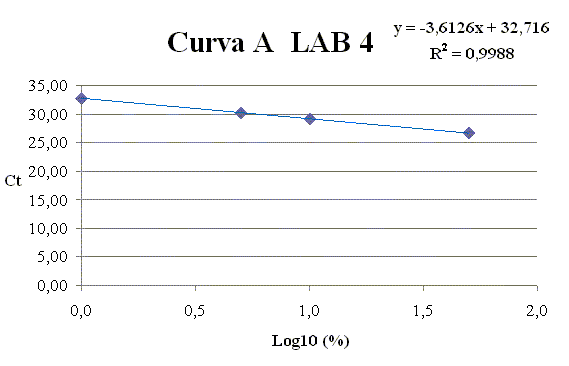
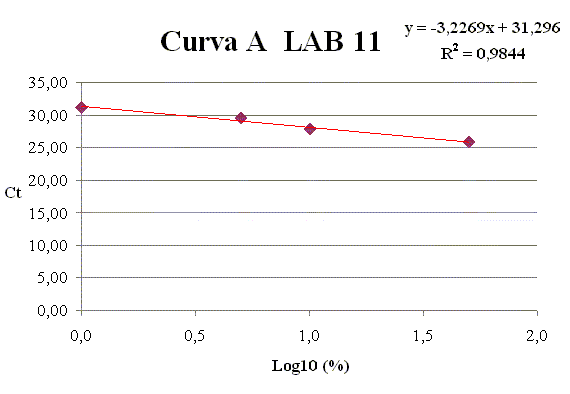
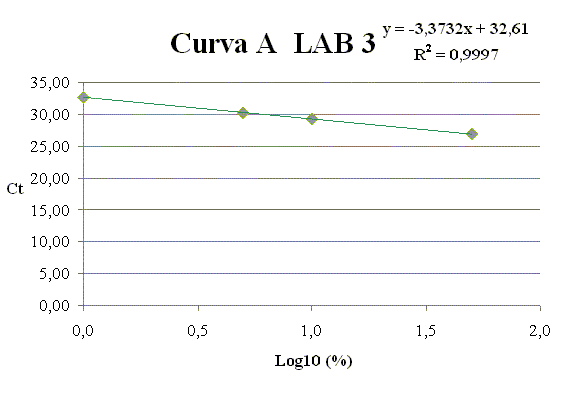
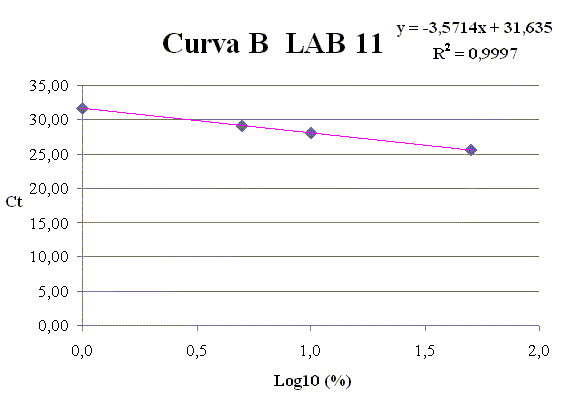
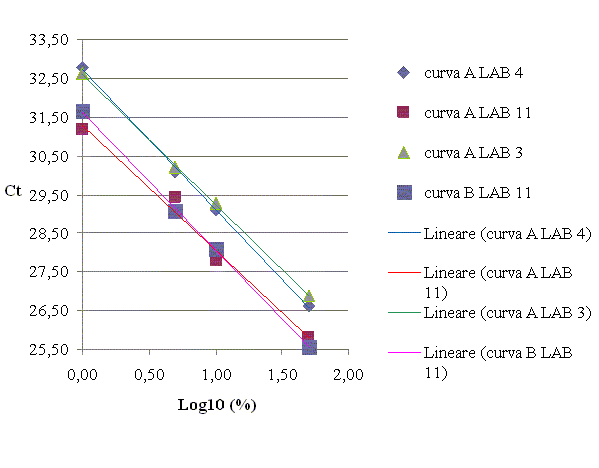
Figura 2. Rette di calibrazione riguardanti le
quattro migliori curve ottenute dai LAB 3, 4, 11.
La curva di calibrazione A, risultata la migliore in termini di slope e R2 per tutti e tre i laboratori coinvolti, è stata quindi ritenuta idonea come materiale di riferimento da impiegare nella quantificazione della soia in matrici complesse.
Conclusioni
Secondo quanto stabilito dalla Direttiva 96/25/CE, nella preparazione o fabbricazione di un prodotto per l'alimentazione animale, non è considerato ingrediente e pertanto non va riportata in etichetta, una materia prima che sia presente in quantità inferiore al 5%. Tuttavia la composizione ed i trattamenti tecnologici associati alla preparazione dei mangimi rendono a volte difficile valutare l' accidentalità della presenza di ingredienti non dichiarati. Va inoltre sottolineato come al momento non siano disponibili in commercio materiali di riferimento che certifichino la quantità del singolo ingrediente presente all'interno di una matrice complessa. Questo lavoro può essere considerato la fase preliminare di un possibile approccio alla risoluzione di tale problema.
A fronte dei risultati ottenuti è stata infatti individuata una retta di calibrazione da impiegare nella quantificazione del singolo ingrediente. Tale retta, allestita con materiali di riferimento preparati in laboratorio, risponde ai parametri di linearità e di efficienza della reazione definiti da ENGL (Reg. 1829/2003 CE). Il metodo Real Time PCR investigato si è dimostrato in grado di rilevare la presenza del singolo ingrediente soia all'interno di una matrice complessa fino a un minimo di 0.1 % (LOD) e di quantificarne la concentrazione fino a un minimo di 1% (LOQ).
Questi dati suggeriscono quindi l'impiego di tale metodo per la ricerca e la quantificazione del singolo ingrediente. Tuttavia sarebbe auspicabile condurre ulteriori indagini per poter definire e disporre di Materiali di Riferimento certificati da impiegare per le matrici e per le concentrazioni investigate in questo lavoro.
Ringraziamenti
Le prove eseguite in questo lavoro sono state condotte nell'ambito del progetto di ricerca corrente finanziato dal Ministero della Salute: "Messa a punto e validazione di un metodo analitico in Real Time PCR per la quantificazione di ingredienti (specie vegetali) in matrici complesse" (RC IZSUM 20/2008).
Bibliografia
Berdal, KG., Holst-Jensen A. (2001). Roundup Ready® soybean event-specific real-time quantitative PCR assay and estimation of the practical detection and quantification limits in GMO analyses. European Food Research and Technology, 213: 432-438.
DIRETTIVA 95/25/CE - circolazione e utilizzazione delle materie prime per mangimi, che modifica le direttive 70/524/CEE, 74/63/CEE, 82/471/CEE, 93/74/CEE e che abroga della direttiva 77/101/CEE.
European Network of GMO Laboratories (ENGL). Definition of Minimun Performance Requirements for Analytical Methods of GMO Testing, (13 October 2008).
Kodama T., Kurosawa Y., Kitta K., Naito S. (2010). Tendency for Interlaboratory Precision in the GMO Analysis Method Based on Real-Time PCR. The Journal of AOAC International, 93 (2): 734-745.
Martinelli L., Benvenuto E., Dalla Costa L., Nobili C. (2007). Manuale di laboratorio per l'analisi di OGM in matrici agroalimentari. ENEA Ente per le Nuove tecnologie l'Energia e l'Ambiente, Roma.
REGOLAMENTO (EC) 1829/2003 sull'immissione sul mercato di alimenti e mangimi contenenti, costituiti o derivati da OGM
REGOLAMENTO (EC) 1830/2003 sulla tracciabilità ed etichettatura di OGM e sulla tracciabilità di alimenti e mangimi ottenuti da OGM.

Validazione di un metodo analitico in Real Time PCR per la quantificazione di ingredienti di specie vegetali in matrici complesse: studio preliminare by Ciarrocchi F. et al., 2012 is licensed under a Creative Commons Attribuzione - Non commerciale 3.0 Italia License.
Permissions beyond the scope of this license may be available at http://indice.spvet.it/adv.html.