Biblioteca Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
Webzine Sanità Pubblica Veterinaria: Numero 60, Giugno 2010 [http://www.spvet.it/] ISSN 1592-1581
Documento reperibile all'indirizzo: http://spvet.it/indice-spv.html#474
Allestimento e validazione di nuove proposte diagnostiche biomolecolari (Macroarrays e Biosensori) per il rilevamento dei principali agenti biologici in Zootecnia - Set up and validation of new molecular diagnostic tools (Macroarrays and biosensors) for detection of the major biological agents in animal husbandry
Biagetti M., Barocci S., Angeletti M., De Montis A.
Abstract. In the present study were set up and validated two chips, the first for the simultaneous detection of virulence factors of E. coli and one for the detection of the main agents of abortion in ruminants. It was also used optical biosensor for the determination of ovine prion protein genotype.
Riassunto. Nella presente ricerca sono stati allestiti e validati due chip, il primo per il rilevamento simultaneo dei fattori di virulenza di E.coli e l'altro per il rilevamento dei principali agenti abortigeni dei ruminanti. Inoltre è stato utilizzato un biosensore ottico per la determinazione del genotipo della proteina prionica ovina.
Introduzione
La presente ricerca si proponeva di allestire e validare metodi diagnostici innovativi biomolecolari (macroarray e biosensori) per il rilevamento di agenti biologici nel campo della zootecnia, inoltre è stata utilizzata una tecnologia basata su un biosensore ottico per la determinazione del genotipo della proteina prionica degli ovini. La ricerca era articolata nelle seguenti attività: A) Preparazione e validazione di sistemi a micropiastra per il rilevamento di fattori di virulenza di ceppi di E.coli e successiva preparazione di un chip dedicato; B) Validazione di un chip per il rilevamento dei principali agenti abortigeni dei ruminanti (proseguimento RC IZSUM 04/04); C) Applicazione di un biosensore per il test di genotipizzazione ovina.
Attività A: Preparazione e validazione di chip dedicato al rilevamento dei fattori di virulenza di ceppi di E. coli.
L'obiettivo finale delle attività nell'ambito del presente progetto era lo sviluppo e la validazione di una piattaforma che consentisse l'amplificazione e la successiva rivelazione di bersagli genici multipli (FIG.1). Tale piattaforma viene applicata per la rilevazione dei fattori di virulenza di E.coli (antigeni fimbriali: F18, K88, K99, F41, 987P; tossine: Sta, Stb, LT, Stx2e).
Attività B: Validazione di un chip per il rilevamento dei principali agenti abortigeni dei ruminanti
(Brucella spp, Toxoplasma gondii, Neospora spp, Clamidia spp, Listeria monocytogenes, Leptospira interrogans)
Attività C: Applicazione di un biosensore per il test di genotipizzazione ovina.
Scopo della presente attività era lo sviluppo di un sistema di genotipizzazione ovina basato sull'impiego di biosensori ottici.
Metodologia seguita per l'attività A e B
Il DNA estratto da ceppi di E.coli è stato amplificato mediante multiplex PCR e i relativi ampliconi sono stati analizzati sia su gel di agarosio sia tramite chip. La lettura del chip e la relativa interpretazione dei pattern è stata eseguita in modo automatico mediante lettore e software dedicati. Sulla base dello stesso principio è stato validato un chip per il rilevamento dei principali agenti abortigeni dei bovini (RCIZSUM04/04).

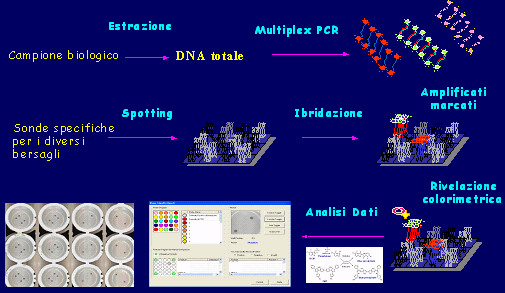
Figura 1. schema del chip per il rilevamento di bersagli genici multipli
Metodologia seguita per l'attività C
Sonde a ssDNA derivatizzate in posizione 5' con gruppi tiolici sono state immobilizzate sulla superficie d'oro della cuvetta del biosensore (FIG.2). Tali sonde sono state disegnate in modo tale da riconoscere le seguenti mutazioni A136V, R154H, Q171R, Q171H.
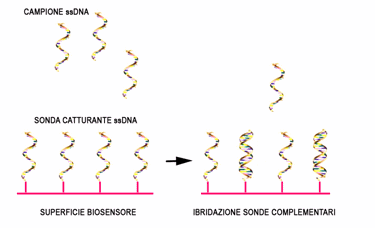
Figura 2. schema di genotipizzazione tramite biosensore
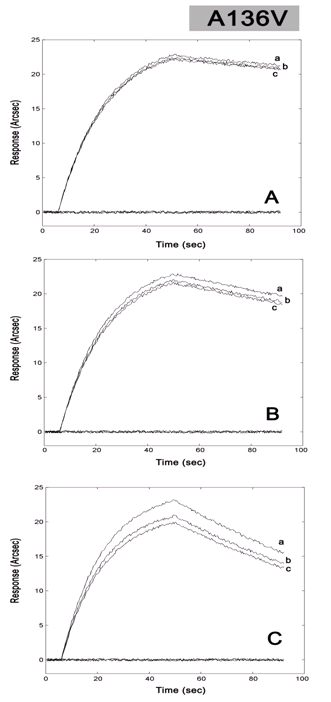
Figura 3. confronto tra curve di legame per ibridazione di sonda specifica per Ala al codone 136 con campioni di genotipi ARQ/VRQ (a), ARQ/ARQ (b) e VRQ/VRQ(c). Esperimenti condotti in soluzione tampone PBS 10mM, NaCl 100mM (A), NaCl 50mM (B) e NaCl 10mM (C).
Risultati delle attività A e B
Dal raffronto dei dati ottenuti tramite lettura visiva su gel con i risultati ottenuti con il chip è emerso che il chip oltre ad aver identificato tutti i fattori di virulenza ha rilevato fattori di patogenicità non visibili su gel. Il chip risulta quindi essere più sensibile ed in grado di svelare anche minime quantità di DNA amplificato non rilevabili su gel. La validazione del chip per agenti abortigeni è stata effettuata su 30 campioni positivi, identificati tramite PCR simplex, e 5 campioni negativi. Dall'analisi dei dati sperimentali emersi, il sistema presenta un'elevata specificità per ciascuno dei target genici identificati; non si osserva infatti alcuna crossibridazione. La specificità è così elevata che l'uso di tale test ha consentito in un campione, risultato positivo in PCR solo per Neospora, di evidenziare una positività anche per Clamidia, confermata tramite sequenziamento.
Risultati dell'attività C
Complessivamente, nonostante le condizioni di elevata stringenza utilizzate, le evidenze sperimentali indicano che le sonde a ssDNA non riescono a conferire alla superficie del biosensore un'adeguata selettività nei confronti di campioni di DNA presentanti diverse varianti alleliche (Fig.3).
Bibliografia
Diringer, H. (1995) Proposed link between transmissible spongiform encephalopathies of man and animals. Lancet 346, 1208-10.
Jeffrey, M., and Gonzalez, L. (2004) Pathology and pathogenesis of bovine spongiform encephalopathy and scrapie. Curr Top Microbiol Immunol 284, 65-97.
Wang R., Minunni M., Tombelli S., Mascini M. A new approach for the detection of DNA sequences in amplified nucleic acids by a surface plasmon resonance biosensor. Biosensor and Bioelectronics (2004) 598-605.
Paton, A. W., and J. C. Paton. 2002. Direct detection and characterization of Shiga toxigenic Escherichia coli by multiplex PCR for stx1 , stx2 , eae, ehxA, and saa. J. Clin. Microbiol. 40:271-274.
Hornes, E., Y. Wasteson, and Ø. Olsvik. 1991. Detection of Escherichia coli heat stable enterotoxin genes in pig stool specimens by an immobilized, colorimetric, nested polymerase chain reaction. J. Clin. Microbiol. 29:2375-2379
Affiliazioni:
M. Biagetti (a), S. Barocci(a), M. Angeletti(b), A.De Montis(c)
(a) Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche, (b) Università degli Studi di Camerino – Facoltà di Scienze e Tecnologie – Scuola di Specializzazione in Biochimica Clinica, (c) BCS Biotech Spa Cagliari






