Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
Webzine Sanità Pubblica Veterinaria: Numero 57, Dicembre 2009 [http://www.spvet.it/] ISSN 1592-1581
Documento reperibile all'indirizzo: http://spvet.it/indice-spv.html#444c
Characterization biochemical and molecular of Escherichia coli isolated from rabbit farms of Central Italy - Caratterizzazione biochimica e molecolare di ceppi di Escherichia coli isolati da allevamenti cunicoli del Centro Italia.
Dettori A., D’Angelo G., Grelloni, V., Mangili P.M., Maresca C., Pezzotti G., Sebastiani C., Magistrali C.F.
Abstract. Escherichia coli has been isolated from healthy and diarrhoeic rabbits in 21 farms in central Italy. The isolates (#111) have been biotyped and tested for antibiotic sensitivity. In addition, the presence of virulence genes (eae, AF/R1, AF/R2) has been investigated through PCR. The biotypes B30 and B28 were mostly identified (53% of the isolates). All the strains showing the gene AF/R2 were also positive for the eae gene demonstrating a potential pathogenicity. On the other hand, there were no isolates with AF/R1 gene, confirming the low frequency of these strains in the Italian farms. No strains were colistin resistance, while common resistance to quinolones, aminoglycosides and sulfonamides was recorded, similarly to what described in other intensively reared animal species.
Riassunto. 111 ceppi di Escherichia coli sono stati isolati in 21 aziende del Centro Italia da conigli con e senza diarrea. Gli isolati sono stati biotipizzati e testati per valutare la sensibilità agli antimicrobici. E' stata valutata inoltre mediante PCR, la presenza dei geni di virulenza (eae, AF/R1, AF/R2). I biotipi B30 e B28 sono stati quelli isolati più frequentemente (53% degli isolati). Tutti i ceppi con AF/R2 presentavano anche il gene eae che dimostrano la potenziale patogenicità del germe. Non è stato isolato il gene AF/R1 confermando la bassa frequenza di isolamento di questa adesina fimbriale nelle aziende italiane. Nessun ceppo è risultato resistente alla colistina mentre sono state evidenziate resistenze a chinoloni, aminoglicosidi e sulfamidici simili ad altre specie animale allevate intensivamente.
Introduzione
Le enteriti sostenute da Escherichia coli sono la causa più frequente di malattia negli allevamenti cunicoli intensivi determinando in questi alti livelli di morbilità e mortalità. I ceppi responsabili della malattia sono definiti REPEC (Rabbit Enteropathogenic Escherichia coli) appartenenti al gruppo AEEC (Attaching Effacing Escherichia coli) (Milon et al, 1999) la cui patogenicità si esplica con l’adesione alla mucosa intestinale dell’ileo distale e del cieco e provoca la distruzione dei microvilli degli enterociti. Questo meccanismo è dovuto alla presenza di un’isola di patogenicità chiamata LEE (Locus of Enterocytes Effacement) all’interno della quale il gene eae codifica per una proteina di membrana, l’intimina. La capacità di aderire agli enterociti e di colonizzare l’intestino oltre che alla produzione di intimina può essere legata anche alla sintesi da parte di E. coli di alcune proteine come le adesine fimbriali AF/R1-AF/R2 (Adesive Factor/Rabbit 1-2). (Dow A.M. et al. 2004)
Scopo di questo studio è stato quello di caratterizzare i ceppi di E. coli isolati da conigli con o senza sintomatologia enterica, individuandone così i principali biotipi, resistotipi, e valutandone la presenza o meno dei geni responsabili della virulenza (eae, AF/R1, AF/R2).
Materiali e metodi
Sono state campionate 21 aziende distribuite nei territori di Umbria, Marche, Lazio e Toscana, 18 delle quali con tipologia di allevamento a “ciclo chiuso” avendo insieme il settore riproduttori e ingrasso e 3 con solo ingrasso.
In ciascuna azienda sono stati prelevati i tamponi rettali da 10 conigli nella fase di post-svezzamento. I tamponi sono stati prelevati in modo casuale.
I tamponi sono stati sottoposti ad esame batteriologico per la ricerca di E.coli mediante semina su Agar McConkey (Oxoid) ed incubati a 37°C ±1°C overnight. Almeno una colonia da ciascun campione è stata isolata su terreno TSA (Trypticase Soy Agar), ed è stata caratterizzata in base a quanto descritto da Carter et al. 1994.
I ceppi isolati sono stati biotipizzati in base allo schema di Camguilhem et Milon (1989) e sottoposti ad agglutinazione rapida su vetrino per il rilievo dell’antigene O103; i ceppi appartenenti a questo sierogruppo sono infatti considerati una importante causa di diarrea nei conigli svezzati (Marchès O. et al, 1999). I geni codificanti per i fattori di patogenicità (eae, AF/R1, AF/R2) sono stati identificati mediante multiplex-PCR (Gannon et al, 1993, Penteado A.S. et al, 2002). E’ stata valutata inoltre la sensibilità agli antimicrobici secondo quanto indicato dalle norme CLSI M31-A2.
Gli antibiotici testati sono stati i seguenti: acido nalidixico (Na), aminosidina (An), apramicina (Apr), colistina (Ct), enrofloxacin (Enr), flumequina (Ub), gentamicina (Cn), kanamicina (K), neomicina (N), sulfametossazolo+trimethoprim (Sxt), tetraciclina (Te).
Risultati
Il numero dei campioni positivi è stato 111 su un totale di 210 campioni testati. In 20 aziende su 21 esaminate è stato isolato almeno un ceppo di E. coli. 2 allevamenti su 21 hanno segnalato la comparsa di diarrea nel post-svezzamento nella settimana precedente il campionamento.
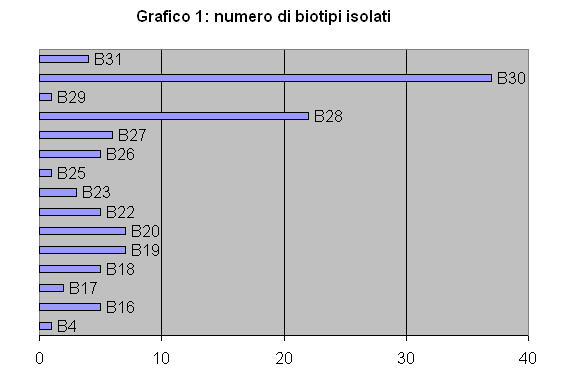
Dei 111 ceppi di E. coli, i dati riguardanti la tipizzazione biochimica sono stati i seguenti: il biotipo B30 è stato isolato in 37/111 (32%); 22/111 (19%) appartenevano al biotipo B28; 7/111 (6%) a B19 e B20; 6/111 (5%) a B27; 5/111 (5%) a B16, B18, B22, B26. 4/111 (4%) a B31; 3/111 (3%) a B23 e 1/111 (1%) a B4, B25, B29 (Grafico 1). 11 ceppi su 111 hanno fornito esito positivo con l’antisiero O 103.
La tipizzazione dei geni codificanti per i fattori di virulenza ha evidenziato la presenza del gene AF/R2 in 19 ceppi, mentre il gene AF/R1 non è mai stato riscontrato; il gene eae è stato isolato in 31/111 ceppi. Tutti i 19 ceppi positivi ad AF/R2 presentavano anche il gene eae ed appartenevano ai biotipi B30 (n=4), B28 (n=8), B22 (n=1), B20 (n=3), B16 (n=2), B4 (n=1).
L’associazione tra l’appartenenza ai diversi biotipi e la presenza di geni codificanti per i diversi fattori di patogenicità, nonché l’antigene O103, relativamente ai biotipi rappresentati da almeno 5 ceppi, è indicata in tabella 1. Un solo ceppo B4 ramnosio negativo è risultato essere positivo per entrambi i geni di virulenza e per l’O103 questo ceppo è stato isolato in un allevamento in cui non si era osservata la presenza di diarrea nel corso della settimana precedente. In tutte e due le aziende con anamnesi positiva per diarrea è stato isolato un ceppo di E.coli eae e AF/R2 positivo. Per quanto riguarda la sensibilità agli antimicrobici, i dati relativi alle percentuali di sensibilità sono indicati in tabella 2.
Il resistotipo più frequente è stato SXTTE (43 ceppi) (sulfametossazolo+ trimethoprim e tetraciclina) seguito dall’associazione apramicina, gentamicina e tetraciclina (10 ceppi) e apramicina, gentamicina. Sulfametossazolo + trimethoprim (8 ceppi). 25 ceppi hanno evidenziato caratteristiche di multiresistenza, ad almeno 5 antibiotici testati. Tra questi 18 ceppi sono risultati resistenti ad almeno un chinolone (acido nalidixico e/o enrofloxacin e/o flumequina), un aminoglicoside (aminosidina e/o apramicina e/o gentamicina e/o kanamicina e/o neomicina) e la tetraciclina.
| Tabella 1: Sono indicati per ogni biotipo i risultati della ricerca dei geni eae, AF/R2 e agglutinazione per O103 |
|---|
| n. biotipo |
eae+ |
eae- |
AF/R2+ |
AF/R2- |
O103+ |
O103- |
| B30 (n=37) |
7 |
30 |
4 |
33 |
1 |
36 |
| B28 (n=22) |
10 |
12 |
8 |
14 |
0 |
22 |
| B19 (n=7) |
1 |
6 |
0 |
7 |
1 |
6 |
| B20 (n=7) |
4 |
3 |
3 |
4 |
0 |
7 |
| B27 (n=6) |
0 |
6 |
0 |
6 |
3 |
3 |
| B16 (n=5) |
3 |
2 |
2 |
3 |
0 |
5 |
| B18 (n=5) |
0 |
5 |
0 |
5 |
2 |
3 |
| B22 (n=5) |
2 |
3 |
1 |
4 |
1 |
4 |
| B26 (n=5) |
3 |
2 |
0 |
5 |
0 |
5 |
| Tabella 2: esito dei test per la valutazione della sensibilità agli antimicrobici |
|---|
| antimicrobico |
S |
% |
R |
% |
I |
% |
| Ac.nalidixico |
76 |
68.5 |
31 |
27.9 |
4 |
3.6 |
| Aminosidina |
96 |
86.5 |
12 |
10.8 |
3 |
2.7 |
| Apramicina |
78 |
70.3 |
31 |
27.9 |
2 |
1.8 |
| Colistina |
109 |
98.2 |
0 |
0.0 |
2 |
1.8 |
| Enrofloxacin |
81 |
73.0 |
7 |
6.3 |
23 |
20.7 |
| Flumequina |
78 |
70.3 |
14 |
12.6 |
19 |
17.1 |
| Gentamicina |
74 |
66.7 |
32 |
28.8 |
5 |
4.5 |
| Kanamicina |
50 |
45.0 |
16 |
14.4 |
45 |
40.5 |
Neomicina
|
34 |
30.6 |
14 |
12.6 |
63 |
56.8 |
Sulfametossazolo+
trimethoprim |
27 |
24.3 |
83 |
74.8 |
1 |
0.9 |
| Tetraciclina |
1 |
0.9 |
109 |
98.2 |
1 |
0.9 |
Conclusioni
I dati derivanti dalle biotipizzazioni dei ceppi di Escherichia coli vedono la predominanza di alcuni biotipi, quali B30 e B28, che insieme raggruppano il 53% degli isolati, e che già altri autori hanno segnalato come prevalenti negli allevamenti cunicoli (Agnoletti et al 2004; Penteado et al 2002). Altri biotipi, quali il B14 e B12, ramnosio negativi, dotati di particolari caratteristiche di patogenicità, non sono stati rilevati nel corso della nostra indagine.
E’ tuttavia importante ricordare che il campionamento effettuato non ha interessato esclusivamente soggetti sintomatici e quindi la frequenza dei biotipi può differire rispetto a quanto osservato in altri lavori, effettuati su ceppi isolati nel corso della attività diagnostica.
In effetti, l’unico ceppo ramnosio negativo isolato nel corso di questo lavoro, appartenente a B4, è risultato positivo per tutti i fattori di patogenicità ricercati. Nel corso di questa indagine, tutti i ceppi positivi per AF/R2 sono risultati positivi anche pereae; inoltre in entrambi gli allevamenti che avevano presentato diarrea nella settimana precedente al campionamento è stato isolato un ceppo con queste caratteristiche. Questa associazione, già osservata da altri autori, è ritenuta infatti fortemente indicativa per l’autonoma capacità patogena del germe (Penteado et al. 2002).
Infine, il gene codificante per AF/R1 non è stato mai riscontrato, confermando la rarità di questa adesina fimbriale, tipica dello stipite RDEC-1, nei ceppi di campo italiani (Agnoletti et al 2004).
Per quanto riguarda i dati di sensibilità agli antimicrobici, essi rilevano buoni risultati per la colistina, nei confronti della quale la resistenza tende a insorgere lentamente. I dati relativi a chinoloni, aminoglicosidi e sulfamidico associato a trimethoprim sono simili a quelli riscontrate in altre specie allevate intensivamente, e testimoniano il diffuso impiego di queste classi di antibiotici nella pratica. Questo dato viene confermato dalla presenza di numerosi stipiti multiresistenti. Essi sottolineano l’importanza di un monitoraggio della resistenza agli antimicrobici in una specie considerata ‘minore’ , ma caratterizzata da un ciclo produttivo breve.
Bibliografia
Agnoletti F., Deotto S., Passera A., Tisato E., Mazzolini E. Diagnosi di colibacillosi nelle sindromi enteriche del coniglio. Coniglicoltura, 2004. (5) 40-41.
Quinn P.J., Carter G.R. Markey B.K., Carter G.R. Clinical Veterinary Microbiology, 1994 Ed. Mosby.
CLSI (exNCCLS) M31-A2 “Performance standards for antimicrobial disk and susceptibility tests for bacteria isolate from animals”. 2002. Second edition. Approved standard.
Crotti S., Grelloni V., Checcarelli S., Cucco L., Mangili P., Magistrali C. Messa a punto di una multiplex PCR (m-PCR) per la ricerca dei geni di patogenicità di E. coli del coniglio. VIII Congresso Nazionale SIDILV Perugia 9-10 novembre 2006 262-263.
Dow M.A., Tòth I., Alexa P., Davies M., Malik A., Oswald E., Nagy B. Predominance of afr2 and ral fimbrial genes related to those encoding the K88 and CS31A fimbrial adhesins in enterophatogenic Escherichia coli isolates from rabbits with postweaning diarrhea in central Europe. J.Clin. Microb. 2005. 1366-1371.
Gannon V.P.J., Rashed M., King R.K., Thomas E.J.G., Detection and characterization of the eae gene of shiga-like toxin-producing Escherichia coli using polymerase chain reaction. J.Clin. Microb. 1993. 31, 1268-1274.
Marchès O., Nougayrède J., Boullier S., Mainil J., Charlier G., Raymond I., Pohl P., Boury M., De Rycke J., Milon A. Oswald E. Role of Tir and intimin in the virulence of rabbit enteropathogenic Escherichia coli serotype O103:H2 Infect Immun. 2000 Apr; 68(4): 2171-82.
Milon A., Oswald E., De Rycke J. Rabbit EPEC: a model for the study of enteropathogenic Escherichia coli. Vet Res. 1999. Mar-Jun;30(2-3):203-19.
Penteado A.S., Ugrinovich L.a., Blanco J., Blanco M., Blanco J.E., Mora A., Andrade J.R.C., Correa S.S., Pestana de Castro A.F. Serobiotypes and virulence genes of Escherichia coli strains isolated from diarrheic and healthy rabbits in Brasil. Vet. Microbiol. 2002. 89, 41-51