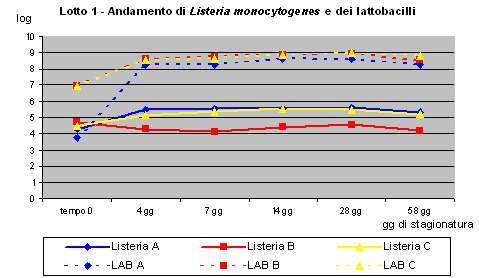
| Tab. 1 - Salami contaminati con L. monocytogenes | ||||||
| Aw | pH | |||||
| A | B | C | A | B | C | |
| 0 | 0.97 | 0.96 | 0.97 | 5.83 | 5.70 | 5.75 |
| 4 gg | 0.95 | 0.96 | 0.96 | 4.83 | 4.82 | 4.80 |
| 7 gg | 0.94 | 0.95 | 0.94 | 4.66 | 4.73 | 4.78 |
| 14 gg | 0.92 | 0.92 | 0.92 | 4.75 | 4.82 | 4.84 |
| 28 gg | 0.89 | 0.90 | 0.89 | 5.00 | 4.68 | 4.86 |
| 58 gg | 0.83 | 0.83 | 0.83 | 5.12 | 4.92 | 4.92 |
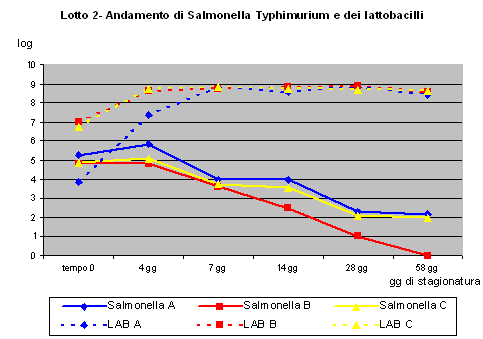
| Tab. 2 - Salami contaminati con S. typhimurium | ||||||
| Aw | pH | |||||
| A | B | C | A | B | C | |
| 0 | 0.97 | 0.97 | 0.97 | 5.79 | 5.75 | 5.78 |
| 4 gg | 0.96 | 0.96 | 0.96 | 4.65 | 4.86 | 4.84 |
| 7 gg | 0.94 | 0.94 | 0.94 | 4.67 | 4.76 | 4.73 |
| 14 gg | 0.93 | 0.93 | 0.93 | 4.66 | 4.73 | 4.70 |
| 28 gg | 0.89 | 0.89 | 0.89 | 4.87 | 4.82 | 4.85 |
| 58 gg | 0.83 | 0.83 | 0.83 | 4.99 | 4.87 | 4.88 |