Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
Webzine Sanità Pubblica Veterinaria: Numero 40, Febbraio 2007 [http://www.spvet.it/]
Documento reperibile all'indirizzo: http://spvet.it/indice-spv.html#363
The antibiotic resistance phenomenon: the IZSUM experience - Il Fenomeno dell'antibiotico-resistenza: esperienze nell'IZSUM
Morgante R. A., (*)Cenci-Goga B. T., Valiani A., Cibotti S., Cenci T.
Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
*Dipartimento di Patologia, Diagnostica e Clinica Veterinaria - Facoltà di Veterinaria Università di Perugia
Abstract: Because of the notable importance of the antibiotic resistance, in the medical and veterinarian field, the IZSUM has performed a research project in order to estimate the entity of the phenomenon.
In this study samples (rectal tampons) delivered to the laboratory in a period of about two years (by 2002 to 2004) have been analyzed in order to appraise the presence of Enterococchi vancomycin-resistant (VRE).
This resistance has been classified in four principal phenotype.
All the Enterococchi VRE stocks have been subsequently subjected to E-test in order to obtain a quantitative evaluation of their resistance towards other antibiotics.
From theis study come out into evidence that, in the largest part of the cases, multiresistence phenomena arise.
This happens because of repeated treatments of many antibiotic in the time.
Nevertheless, from the data, it is evident a strong decrease of the number of isolated VRE stocks.
This is also merit of the European Union that, from April 1997, has forbidden the use, as growth promoters of antibiotic.
Introduzione
Il fenomeno dell'antibiotico resistenza ha assunto, nel corso degli ultimi decenni, sempre più importanza a livello nazionale ed internazionale. Infatti, l'uso di antibiotici a scopo terapeutico e come promotore di crescita, sia in campo umano che veterinario, ha esercitato una pressione selettiva tale da determinare la comparsa di ceppi batterici resistenti a molti dei più comuni antibiotici usati nella pratica clinica.
I principali batteri (spesso multiresistenti) sui quali è focalizzata l'attenzione della maggior parte dei ricercatori internazionali sono Stafilococchi MRSA (Methicillin Resistant Staphylococcus aureus), VRE (Vancomycin-resistant enterococci), Salmonella (DT 104), Escherichia coli, Campylobacter (6) che rappresentano la conseguenza diretta di un uso, talvolta, troppo disinvolto degli antibiotici.
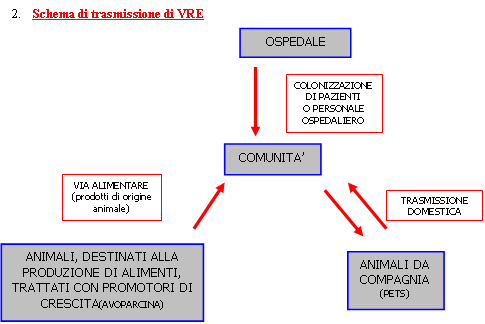
In campo veterinario il fenomeno è conosciuto ormai da decenni e fin dal principio si è cercato di evidenziare eventuali correlazioni con il campo umano. Molte delle ricerche sono state finalizzate ad evidenziare il possibile passaggio di ceppi batterici dotati del carattere dell'antibiotico resistenza, utilizzando gli alimenti di origine animale come veicolo per la trasmissione dagli animali all'uomo (schema 1)(5,13).
Con l'intento di limitare il selezionamento di questi ceppi batterici antibiotico resistenti, l'Unione Europea, a partire dall'aprile del 1997 (EC directive 97/6/EG and Commission regulation EG No. 2821/98 (1), ha vietato l'uso, come promotore di crescita, di alcuni antibiotici (avoparcina, bacitracina, tilosina, spiramicina, virginiamicina) che in alcuni casi vengono impiegati a scopo terapeutico anche nella terapia medica sia umana che veterinaria.
In campo umano il maggior numero di isolamenti è effettuato in ambito ospedaliero; infatti, molte infezioni in cui sono coinvolti questi ceppi batterici è direttamente imputabile ad infezioni nosocomiali (uso di cateteri endovenosi, superfici o materiali non sanificate, personale ospedaliero o ospedalizzato che funge da portatore attivo o passivo).
Ciò si traduce in termini sociali ed economici in un incremento della morbilità e mortalità dei pazienti sottoposti per lungo tempo a trattamenti antibiotici oltre che in un aumento della spesa pubblica a causa della maggiore durata del ricovero ospedaliero, associato ad un maggior numero di trattamenti(2,4).
Premessa
Gli Eterococchi
Gli enterococchi sono batteri che fanno parte della normale flora batterica intestinale sia dell'uomo che degli animali. Generalmente si comportano come commensali, ma in particolari circostanze, si possono comportare come patogeni opportunisti. Sono considerati batteri molto resistenti dato che hanno la capacità di sopravvivere in condizioni ambientali proibitive per la maggior parte degli altri batteri.
Hanno una resistenza genetica nei confronti di alcune classi di antibiotici come aminoglicosidi, cefalosporine e fluorchinoloni. Molti ceppi hanno sviluppato resistenza per alte concentrazioni di aminoglicosidi con una MIC (Minina Concentrazione Inibente) >500 µl o > di 1000 µl, produzione di alti livelli di β-lattamasi e resistenza nei confronti di macrolidi, tetracicline e cloramfenicolo (2,3,5).
Sono comunemente coinvolti in forme setticemiche, mastiti, endocarditi, infezioni delle vie urinarie sia nell'uomo che negli animali domestici. Sono considerati come la seconda più comune causa di infezione e la terza più comune causa di batteriemia in ambito nosocomiale (2).
Vancomicina
La vancomicina è un glicopeptide che agisce a livello del peptidoglicano, inibendone la sintesi.
E' frequentemente impiegata nel trattamento di infezioni causate da MRSA, stafilococchi coagulasi negativi, in meningiti da Streptococcus pneumonie con ridotta sensibilità ai beta-lattamici, così come in pazienti allergici ai beta-lattamici con un'infezione da batteri gram positivi (4).
Molte sono state le correlazioni evidenziate tra l'uso di vancomicina e teicoplanina in campo umano e l'uso di avoparcina in campo zootecnico.
Antibiotico resistenza
Il primo isolamento di VRE risale al 1986 in Francia (4) ed un anno dopo è stato segnalato il primo isolamento documentato negli USA (4). Al 1996 risale il primo isolamento di Stafilococchi resistenti alla vancomicina (VISA)(4).
L'antibiotico resistenza non è una caratteristica esclusiva dei germi patogeni. La flora intestinale può rappresentare un grosso serbatoio per la resistenza batterica e di geni responsabili della resistenza. L'uso di antibiotici induce profondi cambiamenti della nomale flora batterica favorendo la selezione o la colonizzazione di specie batteriche resistenti.
L'ambiente enterico rappresenta il luogo ideale per il trasferimento in vivo di geni dell'antibiotico resistenza (trasferimento orizzontale) tra microrganismi di generi e specie diverse.
Il meccanismo è molto efficiente, infatti, attraverso questa via i germi possono acquisire, mediante un plasmide o un trasposone, un gene o un'intera sequenza di geni correlati alla multiresistenza (8,11,12).
1. Fenotipi di antibiotico resistenza
La resistenza alla vancomicina è stata classificata in 4 principali fenotipi: il così detto fenotipo di resistenza Van-A, che è il più frequente, è inducibile sia con l'uso di teicoplanina (MIC >16 µg/ml) che vancomicina (MIC >128 µg/ml ).
Il fenotipo Van-B mostra livelli variabili di resistenza alla Vancomicina (MIC 4-1024 µg/ml) ma resta sensibile alla teicoplanina (MIC <0,5 µg/ml) ed è inducibile solo dalla Vancomicina. Entrambi i fenotipi Van-A e Van-B sono molto frequentemente associati ad E.faecium ed E.faecalis. La resistenza Van-A e Van-B è generalmente mediata da plasmidi e da trasposoni (Tn 1546).
Il fenotipo di resistenza Van-C (MIC 8-16 µg/ml) è caratterizzato da un basso livello di resistenza alla vancomicina e da sensibilità alla teicoplanina ed è comunemente associato a E. gallinarum ed E. casseliflavus; è legato a geni presenti a livello cromosomiale e non è trasferibile. Il fenotipo di resistenza Van-D è stato evidenziato in un singolo ceppo di E. faecium con resistenza alla vancomicina ed a bassi livelli di teicoplanina. Recentemente, è stato evidenziato anche un altro fenotipo: Van-E con bassi livelli di resistenza alla vancomicina (MIC 16 µg/ml) e sensibile alla teicoplanina (3, 4).
Il carattere dell'antibiotico resistenza può essere associato a geni che in molti casi si rinvengono su elementi genetici mobili come plasmidi e trasposoni e ciò rende il trasferimento di questo carattere intra-inter specifico ed inter-generico (11). Ad aggravare il fenomeno c'è il fatto che nella trasmissione di questi caratteri possono prendere parte germi appartenenti alla normale flora batterica o usati come colture starter (batteri lattici) nei processi tecnologici di produzione di alimenti destinati all'uomo. Infatti, la presenza di geni Van-A e Van-B è stata segnalata anche in batteri diversi dagli Enterococchi: il gene Van-A è stato rinvenuto nei generi lactococcus, orskovia e arcanobacterium, mentre il gene Van-B in streptococcus bovis (5).
2. Schema di trasmissione di VRE
Esperienze dell'IZS UM
Materiali e metodi
Al fine di verificare la presenza di ceppi VRE sono stati considerati i campioni (tamponi rettali) pervenuti al laboratorio in un periodo di circa 2 anni, dal 2002 al 2004. Sono stati adottati criteri random di scelta delle aziende zootecniche regionali (di suini e bovini) ed in tale ambito degli animali da sottoporre a campionatura. I 280 tamponi rettali sia di suini che di bovini sono stati analizzati per la ricerca di Enterococchi vancomicina resistenti.
Isolamento degli Enterococchi
I campioni (5-10 per ogni allevamento) sono stati esaminati utilizzando, in parallelo, i seguenti metodi:
- Semina diretta: VRE agar base + meropenem + gentamicina e VRE agar base + meropenem + vancomicina addizionato rispettivamente di 6g/l di gentamicina e vancomicina, Slanetz- Bartley agar addizionato di vancomicina (6g/l)(Oxoid);
- arricchimento selettivo: VRE brodo + meropenem e seminando, dopo incubazione per 24 ore a 37°C, un'ansata della brodocoltura così ottenuta su VRE + vancomicina, VRE + gentamicina e Slanetz- Bartley + vancomicina (Oxoid).
| Tabella.1: Isolati per specie |
| Specie | Suini | Bovini | Totali |
| E. faecalis | 21 | 40 | 61 |
| E. faecium | 6 | 50 | 56 |
| E. casseliflavus | 0 | 2 | 2 |
| E. gallinarum | 3 | 6 | 9 |
| E. hirae | 1 | 0 | 1 |
| E. spp | 2 | 3 | 5 |
| E. avium | 1 | 1 | 2 |
| E. durans | 2 | 18 | 20 |
| E.spp | 47 | - | 47 |
| Totale | 83 | 120 | 203 |
Le colonie tipiche selezionate, dopo un'incubazione per 24-48 ore a 37°C, sono state sottoposte alle seguenti prove di identificazione: colorazione con metodo di gram, prova della catalasi e test biochimici in micrometodo (API RapidID32Strep, BioMéhrieux) ed i risultati del kit in micrometodo sono stati interpretati con l'ausilio del software Apilab Plus (Vers. 3.3.3, BioMéhrieux). I ceppi, identificati come Enterococchi, per un totale di 203 (Tabella 1) sono stati successivamente sottoposti ad antibiogramma secondo il metodo di Kirby-Bauer, in accordo con gli standard internazionali CLSI (ex NCCLS National Committee for Clinical Laboratori Standards) (14). Gli antibiotici (Oxoid) usati nella prova sono stati: meticillina (MET 5), ticarcillina (TIC 75), cloramfenicolo (C 30), ciprofloxacina (CIP 5), tetraciclina (TE 30), vancomicina (VA 30), gentamicina (CN 10), streptomicina (S 10), kanamicina (K 30), amikacina (AK 30), rifamicina (R 5), tilosina (TY 30).
Tutti i ceppi che hanno manifestato resistenza alla vancomicina al test di Kirby-Bauer (Figura 1), sono stati successivamente sottoposti ad E-test (Figura 2), con lo scopo di ottenere una valutazione anche quantitativa del grado di resistenza degli stessi nei confronti di un più ristretto numero di antibiotici (Grafico 1).
Risultati e discussione
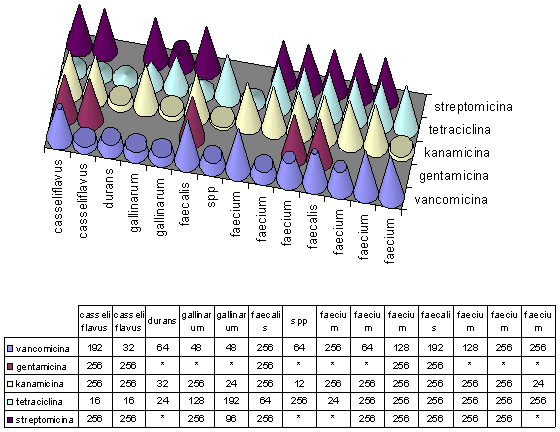 Grafico 1:
Grafico 1: E-Test(µg/ml eseguito nei confronti dei ceppi VRE isolati (*)=sensibile
Analizzando i dati ottenuti (Grafico 1), si possono contare 14 ceppi VRE, di cui:
- 9 dotati di una resistenza alla vancomicina con una MIC >= 64 µ/ml;
- 4 dotati di una resistenza alla vancomicina con MIC >= 256 µg/ml;
- 9 con spiccata multiresistenza (con resistenza ad almeno tre diverse categorie);
- "5 nettamente resistenti alla gentamicina (MIC > di 256 µg/ml).
7 degli 8 ceppi di E. faecium e E. faecalis isolati, ai quali è rivolta una maggiore attenzione a causa del più elevato grado di patogenicità rispetto alle altre specie, hanno mostrato una MIC per la vancomicina alta (MIC >= 128 µg/ml).
La percentuale di isolamento di ceppi VRE è pari a 6,8% del totale dei campioni analizzati, mentre nell'ambito della categoria degli aminoglicosidi, tale percentuale mostra un elevato grado di eterogeneità con valori compresi tra 16,5% per la gentamicina ed 84,2% per l'amikacina. Elevata è anche risultata la resistenza alla meticillina (94%) ed alla tetraciclina (60,0%).
| Tabella.2: Resistenze antibiotici |
| ANTIBIOTICO | Resistenti
|
| N° | % |
| Meticillina | 191 | 94,0 |
| Ticarcillina | 25 | 12,3 |
| Cloramfenicolo | 27 | 13,3 |
| Ciprofloxacina | 28 | 13,7 |
| Tetraciclina | 122 | 60,0 |
| Vancomicina | 14 | 6,8 |
| Gentamicina | 37 | 16,5 |
| Streptomicina | 65 | 32 |
| Kanamicina | 155 | 76,3 |
| Amicacina | 171 | 84,2 |
| Spiramicina | 193 | 95 |
| Rifamicina | 99 | 48,7 |
| Tilosina | 140 | 68,9 |
Conclusioni
In USA, dove l'avoparcina non è stata impiegata come promotore di crescita, il fenotipo di VRE predominante è quello Van-B, la cui selezione è indotta dall'uso di vancomicina e non di avoparcina. Cosa opposta in Europa, dove il fenotipo predominante è Van-A (7) il quale è indotto anche dalla avoparcina, a testimonianza, quindi, di come l'uso di avoparcina in Europa potrebbe aver avuto un ruolo non trascurabile nella comparsa di ceppi VRE (10, 13).
A distanza di alcuni anni dal divieto, imposto a livello europeo, di usare antibiotici come promotori di crescita in campo zootecnico, abbiamo ottenuto risultati concordi con quanto già segnalato da altri Autori che indicano una sensibile diminuzione del numero di ceppi di VRE isolati (7,9).
I ceppi di Enterococchi da noi isolati sono dotati, in molti casi, di multiresistenza; conseguenza evidente, di una serie di pressioni selettive (utilizzo di diversi antibiotici e trattamenti ripetuti nel tempo), esercitate direttamente su di essi o indirettamente su specie batteriche capaci di trasmettere loro il carattere.
Bibliografia
1. F. Haesebrouk,A. Martel, L. Devriese, and A. Decostere:Antibiotic resistance in Gram-positive bacteria isolated from pigs and pork carcasses: possible risks for human health.
2. EARSS Annual report 2001.
3. D. E. Low, N. Keller, A. Bart, and R.N.Jones: Clinical prevalence, antimicrobial susceptibility and geographic resistance patterns of Enterococci: results from the SENTRY antimicrobial surveillance program, 1997-1999. Clinical Infectious Disease, 2001; 32:S133-S145.
4. L.S.Chavers, S.A. Moser, W.H. Benjamin, L.R.Steinhauer, A.M. Smith, C.N. Johnson, E. Funkhouser, L.P. Chavers, A.M Stamm and K.B. Waites: Vancomycin-resistant enterococci: 15 years and counting. Journal of hospital Infection 2003, 53: 159-171.
5. B.E. Murray: Dyversity among multidrug-Resistant Enterococci, EID Vol 4. N° 1.
6. J.R. Bywater and M. W. Casewell: An assesment of the impact of antibiotic resistence in different bacterial species and of the contribution of animal sources to resistance in human infection. Journal of antimicrobial chemoterapy, 2000 46,639-645.
7. M. D. Grosso, A. Caprioli, P. Chinzari, M. C. Fontana, G. Pezzotti, A. Manfrin, E. D. Giannatale, E. Goffredo, and A. Pantosti: Detection and characterization of vancomycine resistant enterococci in farm animals and raw meat products in Italy.Microbial-drug-resistance-Larchmont,N.Y. 2000, 6(4):313-8.
8. E. C. Dancla and J.P.Lafont: Mechanism of resistance to antibiotics in animal and zoonotic pathogens. Veterinary Research 2001 32,(3-4).
9. A. Pantosti, M. del Grosso, S. Tagliabue, A. Macrì, A Caprioli: Decease of vancomycin-resistant enterococci in poultry meat after avoparcin ban. The Lancet, Vol 354, August 28, 1999.
10. K. Gambarotto, MC Ploy, F. Dupron, M. Giangiobbe and F Denis: Occurrence of Vancomycin-Resistant Enterococci in pork and poltry products from a cattle-rearing area of France. Journal of Clinical microbiology, June 2001, p.2354-2355.
11. G.Poli, A. Cocilovo,: Microbiologia ed immunologia veterinaria. Capitolo VII, p. 129-137.
12. The European Agency for the Evaluatio of medicinal Products Veterinary Medicine and Inspections, Veterinary Medicines Evaluation Unit: Antibiotic Resistance in the European Union Associated with Therapeutic use of veterinary Medicines. Report and Qualitative Risk Assessment by the Committee for Veterinary Medicinal products. EMEA/CVMP/342/99- FINAL.
13. B.T. Cenci Goga, M. Karama, P.V. Rossitto, R.A. Morgante end J.S. Cullor: Enterotoxin production by Staphylococcus aureus isolated from mastitic cow. Journal of food protection Vol. 66, No. 9, 2003, pages 1693-1696.
14. CLSI (ex NCCLS) Documents M-31 A2 Performance Standards for Antimicrobial Disk and Dilution Susceptibility Tests for Bacteria Isolated from Animals; Approved Standard.