Biblioteca Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
Sanità Pubblica Veterinaria: Numero 108, Giugno 2018 [http://www.spvet.it/] ISSN 1592-1581
Documento reperibile all'indirizzo: http://spvet.it/indice-spv.html#682
Sorveglianza molecolare sui ceppi di Listeria monocytogenes isolati nell’Umbria e nelle Marche nel 2017
Molecular surveillance on Listeria monocytogenes isolates in Umbria and Marche (Italy) in 2017
Fabrizia Guidi1, Antonietta Gattuso2, Francesca Orecchioni3, Antonella Marziali4, Luciana Gironacci5, Antonella Repetto6, Stefania Scuota1, Donatella Ottaviani1,
Annalisa Petruzzelli1, Stefano Fisichella1, Manuela Lanciotti1, Gianluca Striano1, Barbara Palombo1, Francesco Pomilio7, Giuliana Blasi1
1 Istituto Zooprofilattico Sperimentale Umbria e Marche, "Togo Rosati", Perugia.
2 Dipartimento di Sanità Pubblica, Veterinaria e Sicurezza Alimentare, Istituto Superiore di Sanità, Roma.
3 UOS Microbiologia AOU Ospedali Riuniti, Ancona.
4 UOC Patologia Clinica, INRCA, Ancona.
5 UOC Patologia Clinica, Civitanova Marche, ASUR-AV3, Civitanova Marche.
6 Ospedale Santa Maria della Misericordia, Perugia.
7 Istituto Zooprofilattico Sperimentale dell'Abruzzo e del Molise "G.Caporale", Teramo.
Abstract. Listeria monocytogenes (Lm) is a ubiquitous foodborne pathogen that may cause listeriosis, a severe infection in humans with a very high hospitalisation and case-fatality rate. Lm can grow at refrigeration temperature and low oxygen levels; these properties together with the severity of human listeriosis infections make this pathogen of particular concern for manufacturers of Ready To Eat (RTE) food products. An increase in the occurrence of Listeriosis was observed in Marche region in the period between 2015 and 2016. Characterization of the strains by Pulsed-field gel electrophoresis (PFGE) and Next Generation Sequencing (NGS) allowed identifying a new pattern of L. monocytogenes serotype 1/2a, never isolated in patients before in EU. The results of epidemiological, microbiological and tracing investigations, identified as a likely source of infection a typical sausage known as "coppa di testa", produced by a plant operating in the Region. Aafter this outbreak, the surveillance activity was deemed through characterization of human, environmental and food strains isolated in the Umbria and Marche regions. The aim of this study is to report the results coming from the activity carried out in the year 2017 by the IZSUM in collaboration with the National Reference Laboratory for L. monocytogenes (IZSAM).
Riassunto. Listeria monocytogenes (Lm) è l'agente eziologico della listeriosi, una zoonosi alimentare, che pur avendo bassa frequenza, è quella associata al tasso più alto di ospedalizzazione (97,7% dei casi) e di mortalità (16.2%) (EFSA 2017). L'ubiquitarietà e la spiccata capacità di crescere o sopravvivere in ambiente refrigerato rendono la Lm uno tra gli agenti patogeni più importanti nell'ambito della produzione alimentare, in particolare negli alimenti Ready To Eat (RTE). Nel biennio 2015-2016, nella Regione Marche è stato evidenziato, attraverso il sistema di sorveglianza delle malattie infettive, un aumento dei casi di Listeriosi umana. La Pulsed Field Gel Electrophoresis (PFGE) dei ceppi isolati dai pazienti ha permesso di evidenziare un cluster di infezione, confermato da un ulteriore caratterizzazione mediante Next Generation Sequencing (NGS). Le indagini epidemiologiche e microbiologiche effettuate, hanno individuato come veicolo verosimile, un tipico salume meglio noto come "coppa di testa" prodotto da uno stabilimento operante nella Regione. In seguito a questo episodio si è ritenuto opportuno consolidare l'attività di sorveglianza molecolare mediante attività di caratterizzazione dei ceppi umani, ambientali e alimentari isolati nel territorio di competenza dell'IZSUM. Scopo del presente lavoro è quello di descrivere ed analizzare dal punto di vista epidemiologico i dati ottenuti dall'attività svolta nell'anno 2017, dall'IZSUM in collaborazione con il Laboratorio Nazionale di Riferimento per L. monocytogenes (IZSAM) e l'Istituto Superiore di Sanità (ISS).
Introduzione
Listeria monocytogenes (Lm) è l'agente eziologico della listeriosi, una patologia che colpisce uomo ed animali e che si trasmette principalmente attraverso il consumo di cibo contaminato. L'ubiquitarietà e la spiccata capacità di moltiplicarsi e/o sopravvivere in ambiente refrigerato rendono Lm uno tra gli agenti patogeni più importanti nell'ambito della produzione alimentare.
In particolare, gli alimenti Ready To Eat (RTE), considerate le caratteristiche fisico-chimiche e le modalità di conservazione, costituiscono generalmente un buon substrato per la moltiplicazione di Lm (Montero et al. 2015) e sono pertanto i principali veicoli.
Nelle persone adulte e in buono stato di salute, le infezioni da Lm possono manifestarsi con sintomi simil-influenzali, come nausea, vomito e diarrea, mentre le conseguenze sono decisamente più gravi nelle forme invasive che si manifestano più di frequente nei soggetti con problemi di salute; tra questi rientrano gli immunodepressi, gli anziani, i malati di tumore e i bambini nei quali l'infezione provoca setticemia e meningite, anche le donne in gravidanza sono a rischio in quanto l'infezione può provocare aborto e natimortalità (Di Pinto et al. 2010).
Tra tutte le zoonosi alimentari, la listeriosi, pur avendo bassa frequenza, è quella associata al tasso più alto di ospedalizzazione (97,7% dei casi) e di mortalità (16,2%) (EFSA 2017).
Nella Unione Europea (EU) le infezioni da Lm sono oggetto di sorveglianza in sanità pubblica nell'ambito del programma Foodborne and Waterborne Diseases (FWD) dell'European Centre for Diseases Prevention and Control (ECDC) attraverso il sistema di sorveglianza europeo TESSy (The European Surveillance System) case-based (sorveglianza dei casi clinici di malattia) e TESSy isolate-based (sorveglianza molecolare sugli isolati di Lm) in conformità alla definizione di caso per la listeriosi (Decisione CE 28 Aprile 2008).
La sorveglianza si focalizza sulle forme invasive gravi che si manifestano, per lo più, come setticemia, meningite o aborti. Gli Stati Membri dell'Unione Europea e i paesi dell'Area Economica Europea (Islanda, Liechtenstein, Norvegia) devono comunicare i casi di malattia in conformità alla Decisione n.1082/2013/UE che stabilisce norme in materia di sorveglianza epidemiologica, monitoraggio, allarme rapido e lotta contro le gravi minacce per la salute a carattere transfrontaliero. Il Regolamento CE No 2073/2005 e s.m.i., stabilisce i criteri microbiologici di sicurezza alimentare per Lm negli alimenti.
Dei 13 diversi sierotipi di Lm conosciuti, il sierotipo 1/2a, l'1/2b e il 4b provocano circa il 95% dei casi di listeriosi (Chen et al. 2017). In particolare, i sierotipi 1/2a, 1/2b e 1/2c, sono quelli più frequentemente isolati dagli alimenti, mentre il 4b è quello più spesso associato a focolai epidemici (Montero et al. 2015).
Solo nel 2016, in Europa sono stati confermati 2536 casi di listeriosi, con un trend statisticamente in aumento dal 2008 in ben 12 Stati Membri (MS). Nello specifico, l'Italia è tra i 6 MS che hanno riportato l'aumento più consistente (EFSA 2017).
Nel caso della listeriosi umana, nessuna attività di sorveglianza o indagine epidemiologica può essere considerata completa ed esaustiva senza l'utilizzo di un approccio molecolare volto alla caratterizzazione dei ceppi isolati e all'individuazione di correlazioni epidemiologiche tra questi. In particolare, nel caso di Lm sono utilizzati la sierotipizzazione, la Pulsed-Field Gel Electrophoresis (PFGE) e la Multi Locus Sequence Typing (MLST). Tuttavia, i notevoli passi avanti fatti nell'uso del sequenziamento dell'intero genoma dei batteri (Next Generation Sequencing- NGS) in termini di automatizzazione ed abbassamento dei costi, nonché di analisi bioinformatica dei dati, hanno reso questa tecnica sempre più diffusa e alla portata non solo dei centri laboratoristici più avanzati (Chen et al., 2017). La possibilità di ottenere in tempi rapidi la sequenza di interi genomi, ha rivoluzionato la ricerca applicata fornendo uno strumento di caratterizzazione altamente discriminante da impiegare negli studi epidemiologici (Pettengill et al. 2014). Questo approccio, sempre più diffuso, rappresenta la prospettiva futura di questo settore (Hyden et al. 2016).
In accordo con i dati europei e italiani di prevalenza della listeriosi umana, nel biennio 2015-2016, anche nella Regione Marche è stato evidenziato, attraverso il sistema di sorveglianza delle malattie infettive, un aumento dei casi di Listeriosi umana. La PFGE dei ceppi isolati dai pazienti ha permesso di evidenziare un cluster di infezione e le indagini epidemiologiche e microbiologiche effettuate, hanno individuato come veicolo verosimile, la coppa di testa, un salume tipico prodotto da uno stabilimento operante nella Regione Marche. Il focolaio è stato ampiamente descritto in un report della Regione redatto ad 2016, ma le attività di sorveglianza sono sempre attive, infatti lo scopo del presente lavoro è di descrivere l'attività di sorveglianza molecolare svolta nelle regioni Marche e Umbria nel corso del 2017, mediante la caratterizzazione dei ceppi umani, ambientali e alimentari.
Materiali e Metodi
Raccolta dei ceppi
Tutti i ceppi di Lm isolati presso i laboratori dell'Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche (IZSUM), da alimenti e tamponi ambientali prelevati nell'ambito dell'attività di campionamento dei Servizi dei Dipartimenti di Prevenzione dell'Azienda Sanitaria Unica regionale (ASUR) (Piano Alimenti Umbria e Marche, controlli su richiesta UVAC e altre attività di controllo Ufficiale non pianificate) e i ceppi clinici isolati dai laboratori delle Aziende Sanitarie Ospedaliere delle regioni Umbria e Marche e dalle Unità Operative Complesse di Patologia clinica delle Aree Vaste dell'ASUR, sono stati inviati al Laboratorio Controllo Alimenti di Fermo (LCA10) dell'IZSUM.
Nel caso di alimenti e tamponi ambientali, spesso sono stati isolati fino a 5 ceppi per singola unità campionaria/spugna. Tutti i ceppi disponibili sono stati inviati al Laboratorio Nazionale di Riferimento per Lm (LNR) presso l'IZS Abruzzo e Molise avente sede a Teramo.
Gli isolati clinici sono stati inviati anche all'Istituto Superiore di Sanità (ISS) in quanto Operational Contact Point microbiologico per Lm nel network FWD-ECDC in Italia.
Le informazioni relative ai ceppi sono state inserite nel SEAP, il Sistema Informativo per la Sorveglianza epidemiologica agenti patogeni di origine alimentare, gestito dal LNR per conto del Ministero della Salute.
Identificazione molecolare degli isolati
L'appartenenza degli isolati alla specie Lm, ove necessario, è stata confermata dal LCA10 mediante una PCR end point in grado di rilevare il gene specie hylA, specifico per Lm (E. Thomas et al. 1991). L'estrazione del DNA da amplificare con questo protocollo è stata effettuata incubando a 99°C±2°C per 10 minuti e poi in ghiaccio per 5 minuti, una sospensione ottenuta stemperando quattro colonie isolate da Agar Sangue in 100µL di H2O nucleasi free. La sospensione è stata poi centrifugata a 12000 rpm per 5 minuti recuperando il surnatante. La mix di reazione è stata adattata ad un volume finale di 25µL (20 µL di mix + 5µL di DNA) contenente 1,5 mM di MgCl2, 0,1 mM di dNTPs, 0,5 µM di ciascun primer ed 1U di GoTaq® G2 Flexi DNA Polymerase (Promega, Madison, Wisconsin). Il ciclo termico è stato impostato come segue: pre-attivazione della Taq polimerasi a 95°C per 60s, 40 cicli a 95°C per 15 s, 55°C per 2s e 72°C per 30s, ed un'estensione finale a 72°C per 4 min. La sequenza dei primer utilizzati, le dimensioni dell'amplificato atteso e il relativo riferimento bibliografico sono riportati in Tabella 1.

Table 1. PCR for the identification of
L. monocytogenes strains: target gene, name of the primer, expected amplified and relative bibliography
Per ogni sessione analitica, oltre ai campioni, sono stati saggiati anche un controllo negativo ed uno positivo di estrazione ed un controllo positivo ed uno negativo di amplificazione. Per l'allestimento dei controlli positivi è stato utilizzato il ceppo di Lm ATCC 13932. I prodotti della PCR sono stati visualizzati elettroforeticamente su gel di agarosio all'1,5% in Tris Borato EDTA (TBE) 1X mediante UVIsave Gel Documentation System (UVITEC Cambridge) e dopo colorazione con intercalante Midori Green Advance DNA Stain secondo le specifiche del fornitore (NIPPON Genetics EUROPE GmbH).
Caratterizzazione degli isolati
Determinazione del sierogruppo e del sierotipo
Il sierogruppo è stato identificato da LCA10 mediante Multiplex PCR, acquisendo il metodo in uso presso l'European Union Reference Laboratory for Listeria monocytogenes (ANSES, EURL for Listeria monocytogenes Rev 004:2013) che prevede l'estrazione del DNA genomico in InstaGeneTM Matrix (BioRad, Hercules, California), reagente a base di Chelex al 6% ed il seguente ciclo termico: 94°C per 10min, 35 cicli a 94°C per 40s, 53°C per 90s e 72°C per 90s con un estensione finale a 72°C per 10 min. Il volume finale di reazione è stato adattato a 25µL (23 µL di mix + 2µL di DNA), impiegando il kit commerciale QIAGEN® multiplex-PCR kit e aggiungendo le diverse coppie di primer ad una concentrazione finale di 0,4 µM per LMO0737, LMO1118, ORF2110 e ORF2819, 0,2 µM per LIP e 0,1 µM per PRS. La sequenza dei primer, le dimensioni degli amplificati attesi ed i relativi riferimenti bibliografici sono riportati in Tabella 2.

Table 2. Multiplex PCR for the serogroup identification of
L. monocytogenes strains: target gene, name of the primer, expected amplified and relative bibliography
Per ogni sessione analitica sono stati saggiati anche un controllo negativo ed uno positivo di estrazione, un controllo negativo di amplificazione ed uno positivo per ciascun sierogruppo. I materiali di riferimento utilizzati per l'allestimento dei controlli positivi sono di seguito riportati: ANSES.OOEB248LM (IIa; 1/2 a), ANSES.OOEB249LM (IIb; 1/2 b), ANSES.OOEB250LM(IIc; 1/2 c), ANSES.OOEB254LM (IVa; 4a), ANSES.OOEB256LM (IVb; 4b). I prodotti delle PCR sono stati visualizzati elettroforeticamente su gel di agarosio al 2% TBE 1X e l'interpretazione dei risultati è stata effettuata secondo lo schema riportato in Tabella 3.
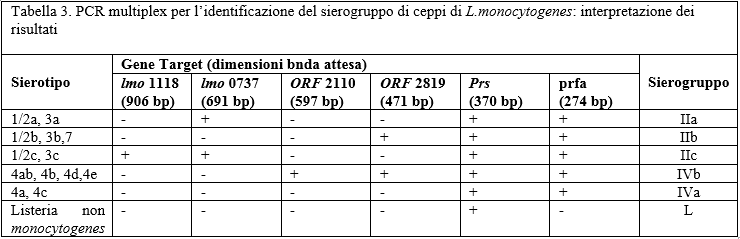
Table 3. Multiplex PCR for the identification of
L. monocytogenesstrains serogroup: interpretation of results
La determinazione del sierotipo degli isolati, che prevede l'identificazione sierologica del profilo antigenico flagellare (H) e di quello somatico (O) di Lm, è stata eseguita dal LNR (IZSAM) secondo il metodo US/FDA Bacteriological Analytical Manual (Bennett et al., 2009), utilizzando sieri commerciali per gli antigeni somatici "O" e flagellari "H" (Denka Seiken CO. LTD, Giappone). Il sierotipo è stato espresso come combinazione degli antigeni del gruppo O e del tipo H.
Tipizzazione molecolare mediante PFGE
La caratterizzazione molecolare dei ceppi mediante PFGE è stata eseguita dal LNR e, per i ceppi clinici anche dall'ISS, secondo il protocollo adottato da PulseNet (CDC, 2013), che prevede come enzimi di restrizione AscI e ApaI e come standard il ceppo Salmonella sierotipo Braenderup (H9812). Le sospensioni batteriche sono state incluse in agarosio, lisate, lavate e digerite con gli enzimi di restrizione.
I campioni in seguito sono stati sottoposti a elettroforesi in agarosio Seakem Gold 1% (Lonza, USA) nel sistema Chef Mapper XA (BioRad Inc, USA) a 6 V/cm con tempo di pulso da 4 sec a 40 sec per 21 ore.
I profili di macrorestrizione sono stati analizzati con il software BioNumerics versione 7.5 (Applied Maths, Belgio) e le similarità state calcolate usando il coefficiente di Dice ed impostando l'ottimizzazione e la tolleranza all'1% per entrambi gli enzimi. Successivamente, il software è stato utilizzato per effettuare la clusterizzazione e la costruzione dei dendrogrammi con il metodo UPGMA (Unweighted Pair Group Method using Arithmetic averages).
Next Generation Sequencing (NGS)
Le analisi di Sequenziamento sui ceppi di Lm, sono state eseguite, qualora ritenuto opportuno, dal LNR, presso il quale è attivo il recente "Centro di Referenza Nazionale per Sequenze Genomiche di Microrganismi Patogeni: Banca Dati e Analisi di Bioinformatica" (CEREM). L'intero genoma batterico è stato sequenziato (Whole-Genome Sequencing-WGS) mediante la tecnologia Solexa-Illumina, utilizzando il Nextera® DNA Library Preparation kit per la preparazione delle librerie e il NextSeq® 500 Illumina® come sistema di sequenziamento.
Il trasferimento dei dati, finalizzato all'analisi bioinformatica, è stato eseguito mediante un sistema interno sviluppato dal CEREM che prevede delle pipeline specifiche per ogni tipologia di analisi.
Consultazione banca dati nazionale
Al fine di individuare eventuali omologie tra i ceppi isolati nel 2017 nell'Umbria e nelle Marche e quelli circolanti negli anni precedenti o in altri territori, è stato consultato il database del LNR contenente i profili PFGE, i Sequence Type (ST) derivanti dall'MLST e le varie informazioni relative ai ceppi di Lm isolati sul territorio nazionale.
Risultati e Discussione
Nel periodo compreso tra il 1 Gennaio e il 31 Dicembre 2017 sono stati collezionati, esaminati e gestiti 158 ceppi di Lm nell'ambito dell'attività diagnostica dell'IZSUM relativa al Controllo Ufficiale Umbria-Marche e al monitoraggio export Paesi terzi Marche. Sette sono stati i ceppi da casi clinici umani, quattro verificatisi nelle Marche e tre nell'Umbria e un ceppo è stato isolato da un agnello. La distribuzione dei ceppi per tipologia di matrice è riportata nella Tabella 4.
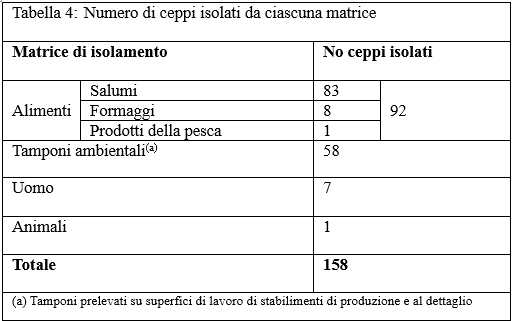
border="0">
Table 4. Number of strains isolated from each matrix
La conferma di identificazione di specie mediante PCR è stata effettuata su 73 isolati e tutti hanno dato esito positivo. Il sierogruppo è stato determinato dal LCA10 per tutti i 158 ceppi con risultati coerenti con quelli ottenuti dal LNR per il sierotipo.
La sierotipizzazione è stata svolta dal LNR per tutti i ceppi provenienti da casi umani di listeriosi, mentre per quelli isolati da alimento e da tamponi ambientali è stato adottato un criterio di selezione.
In particolare, tra tutti gli isolati provenienti dallo stesso campione, è stato caratterizzato almeno un rappresentante per sierogruppo. Nel complesso i ceppi sierotipizzati sono stati 75, tutti riconducibili a quattro sierotipi principali: l'1/2c (47%), l'1/2a (32%), l'1/2b (12%) e il 4b (9%). I sierotipi circolanti sul territorio sono gli stessi individuati in precedenti studi condotti su isolati clinici alimentari ed ambientali, a livello nazionale (Gianfranceschi et al. 2009). La ripartizione per matrice è rappresentata in Tabella 5. Solo i sierotipi 1/2a, 1/2b e 4b sono stati associati a casi umani di listeriosi, mentre tutti i ceppi 1/2c provenivano da alimenti.
Tra gli isolati alimentari sono risultati più frequenti i sierotipi 1/2c e 1/2a, come osservato da Gianfranceschi et al. (2009), mentre tra quelli ambientali sono pressoché ugualmente rappresentati l'1/2a, il 4b e l'1/2b. Per quanto riguarda i ceppi derivanti da casi umani di listeriosi, tra di essi i sierotipi risultati più frequenti sono stati l'1/2a e l'1/2b, mentre l'unico isolato animale era 4b. Considerando l'associazione tra sierotipi e tipologia di alimento, l'1/2a è risultato più frequente nei salumi (13 pari all'81%) come osservato da Gianfranceschi et al. (2009), mentre l'1/2b è stato riscontrato solo nei formaggi (2 isolati) e nell'unico prodotto della pesca (1 isolato); il sierotipo 1/2c, poi, è stato rinvenuto esclusivamente nei salumi con ben 35 isolati, mostrando, coerentemente con quanto riportato da Gianfranceschi et al. (2009), una spiccata associazione con questa tipologia di alimenti.
Questo spiegherebbe anche il motivo per cui il sierotipo 1/2c è risultato in assoluto quello più frequentemente isolato dagli alimenti, considerando l'elevato numero di salumi analizzati rispetto alle altre matrici. Anche l'unico ceppo alimentare 4b, infine, è stato isolato da un salume.
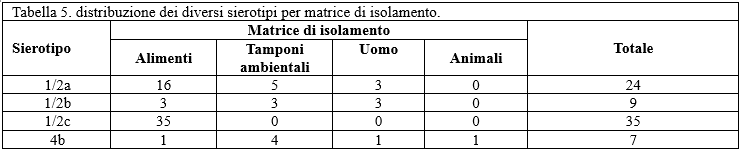
Table 5. Distribution of serotypes by isolation matrix
La PFGE, è stata eseguita dal LNR e dall'ISS per tutti i ceppi clinici, mentre per quelli isolati da alimento e da tamponi ambientali il LNR ha adottato un criterio di selezione. In particolare, tra gli isolati provenienti dallo stesso campione, è stato tipizzato almeno un rappresentante per sierogruppo.
I ceppi tipizzati sono stati in tutto 72 di cui 48 isolati da alimenti, 16 da superfici di lavoro prelevati in stabilimenti di produzione e trasformazione, 7 da casi umani di listeriosi e uno da animale.
L'analisi di macro-restrizione ha generato 28 profili con l'enzima ApaI, 26 con AscI e 30 profili combinati (Tabella 6). La popolazione batterica circolante sul territorio si è rivelata pertanto piuttosto variegata.
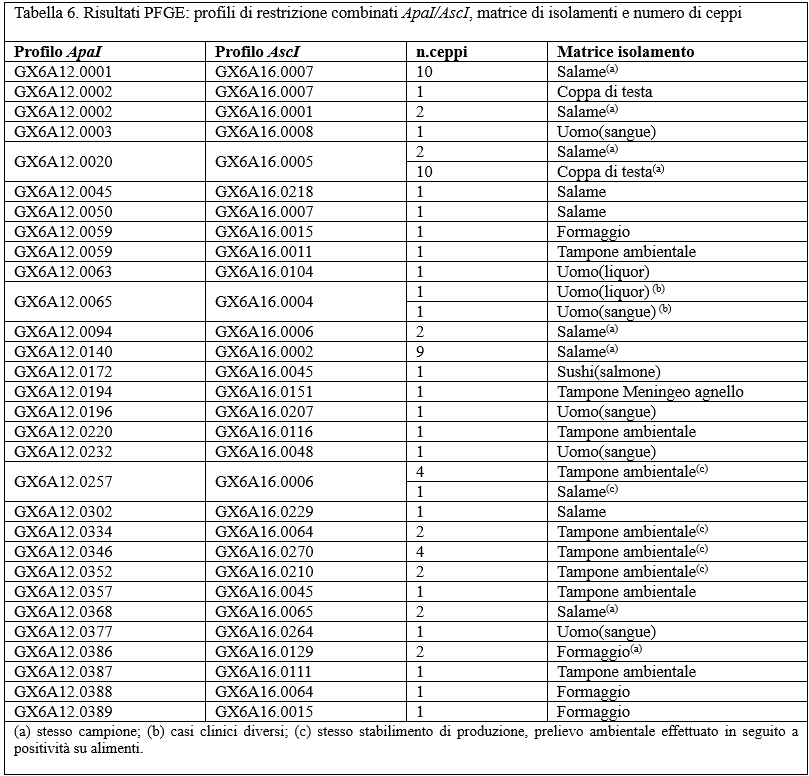
Table 6. PFGE results: combined restriction profiles ApaI / AscI, matrix of isolates and number of strains
Come si evince dalla Tabella 6, nella maggior parte dei casi, i ceppi aventi lo stesso profilo ApaI/AscI sono stati isolati dallo stesso campione di alimento o da tamponi effettuati presso il medesimo stabilimento.
Il profilo GX6A12.0257/GX6A16.0006, isolato da un campione di Salame risultato positivo per Lm è stato successivamente rinvenuto anche nello stabilimento di produzione, nel corso dei prelievi eseguiti in seguito alla positività dell'alimento. Questo suggerisce la possibilità che si trattasse di un ceppo residente nell'impianto di lavorazione.
Il profilo combinato GX6A12.0065/GX6A16.0004, è stato riscontrato in due ceppi clinici isolati da pazienti diversi, i due casi clinici si sono verificati nel mese di Luglio e nella provincia di Ancona. La possibilità che vi fosse una fonte di infezione comune ai due casi non ha trovato conferma nei risultati delle indagini epidemiologiche e i successivi approfondimenti diagnostici mediante NGS non hanno confermato l'indistinguibilità dei ceppi, escludendo l'ipotesi di un focolaio.
Il numero massimo di profili diversi, rinvenuti all'interno di singoli campioni è stato 4 nel caso degli alimenti e 2 per i tamponi. I due alimenti che hanno mostrato una maggiore eterogeneità a livello di popolazione batterica sono stati un formaggio e un salame. La presenza di 4 cloni differenti di Lm sottolinea l'importanza di tipizzare più ceppi isolati dallo stesso campione.
I profili PFGE dei ceppi isolati dai casi clinici di listeriosi, sono stati inseriti nel database dell'LNR al fine di individuare eventuali correlazioni con altri ceppi isolati sul territorio nazionale. Cinque dei sei differenti profili di macrorestrizione ApaI /AscI sono infatti risultati indistinguibili con i profili combinati di ceppi isolati a livello nazionale da varie matrici (Figure 1, 2, 3, 4 e 5).
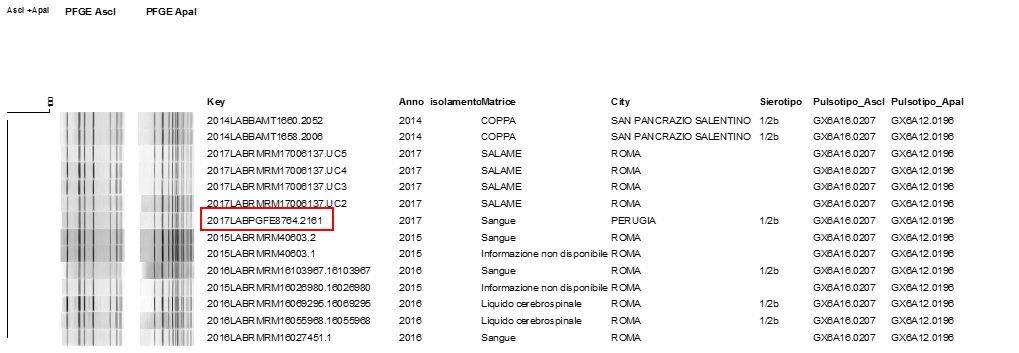
Figura 1. Consultazione del database nazionale LNR: profili PFGE combinati ApaI /AscI GX6A12.0198/GX6A16.0207 di ceppi, matrice di isolamento e luogo di prelievo del campione. Il ceppo clinico è stato evidenziato in rosso
Figure 1. Consultation of the national LNR database: Combined PFGE profiles ApaI / AscI GX6A12.0198 / GX6A16.0207 of strains,
insulation matrix and sampling location. The clinical strain was highlighted in red
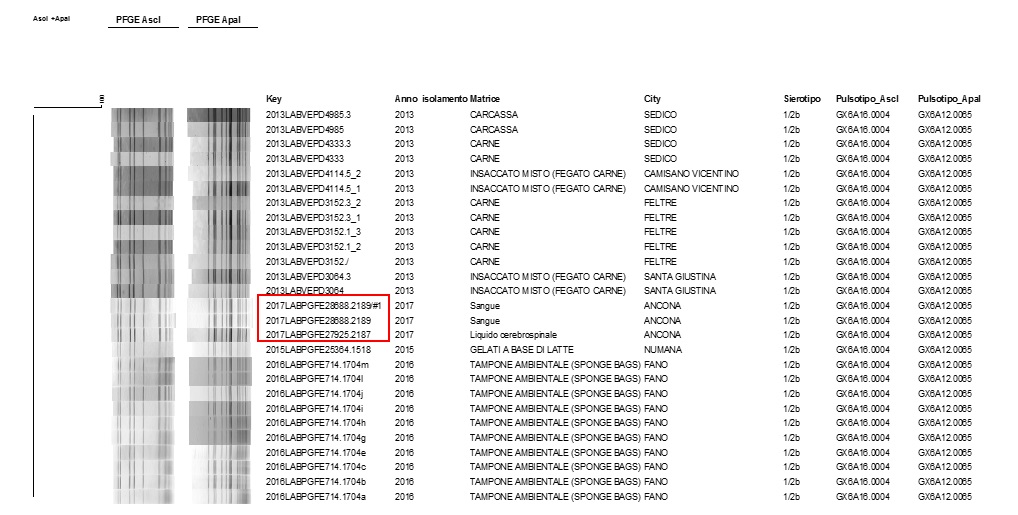
Figura 2. Consultazione del database nazionale LNR: profili PFGE combinati ApaI /AscI GX6A12.0065/GX6A16.0004 di ceppi,
matrice di isolamento e luogo di prelievo del campione. I ceppi clinici sono stati evidenziati in rosso
(il ceppo 2189 è presente in doppio perché per questo ceppo la PFGE è stata ripetuta)
Figure 2. Consultation of the national LNR database: combined PFGE profiles ApaI / AscI GX6A12.0065 / GX6A16.0004 of strains,
insulation matrix and sampling location.
The clinical strains have been highlighted in red
(the 2189 strain is double because for this strain the PFGE has been repeated)
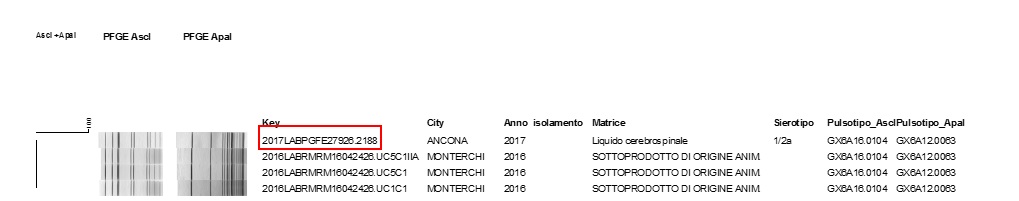
Figura 3. Consultazione del database nazionale LNR: profili PFGE combinati ApaI /AscI GX6A12.0063/GX6A16.0104 di ceppi,
matrice di isolamento e luogo di prelievo del campione. Il ceppo clinico è stato evidenziato in rosso
Figure 3. Consultation of the national LNR database: combined PFGE profiles ApaI / AscI GX6A12.0063 / GX6A16.0104 of strains,
insulation matrix and sampling location. The clinical strain was highlighted in red
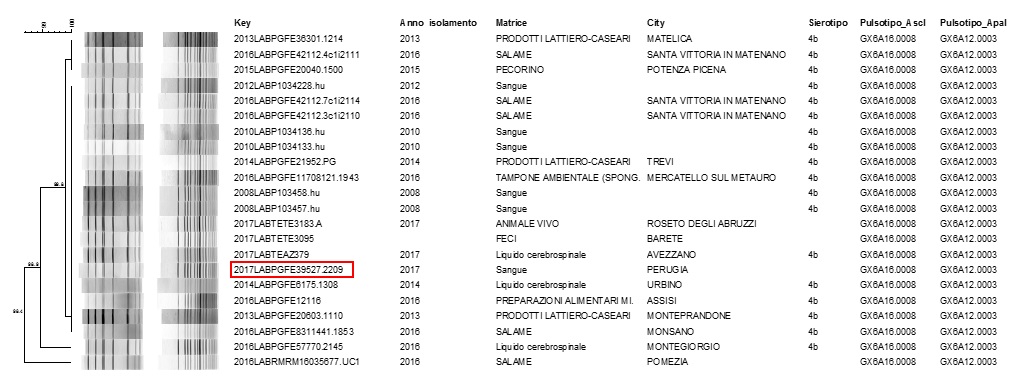
Figura 4. Consultazione del database nazionale LNR: profili PFGE combinati ApaI /AscI GX6A12.003/GX6A16.0008 di ceppi,
matrice di isolamento e luogo di prelievo del campione. Il ceppo clinico è stato evidenziato in rosso
Figure 4. Consultation of the national LNR database: combined PFGE profiles ApaI / AscI GX6A12.003 / GX6A16.0008 of strains,
insulation matrix and sampling location. The clinical strain was highlighted in red

Figura 5. Consultazione del database nazionale LNR: profili PFGE combinati ApaI /AscI GX6A12.0232/GX6A16.0048 di ceppi,
matrice di isolamento e luogo di prelievo del campione. Il ceppo clinico è stato evidenziato in rosso
Figure 5. Consultation of the national LNR database: combined PFGE profiles ApaI / AscI GX6A12.0232 / GX6A16.0048 of strains,
insulation matrix and sampling location. The clinical strain was highlighted in red
I dati epidemiologici e il sierotipo dei ceppi isolati dai casi clinici di listeriosi sono stati inoltre inviati all'European Centre for Disease Prevention and Control (ECDC) attraverso l'European Surveillance System (TESSy) mentre i profili PFGE sono stati inseriti nel database dell'ECDC attraverso l'applicativo web: https://tessy.ecdc.europa.eu"
Conclusioni
Il sistema di gestione dei ceppi di Lm attuato nella Regione Marche e operativo anche per la Regione Umbria rappresenta il consolidamento del flusso operativo implementato durante il focolaio di listeriosi verificatosi nelle Marche tra il 2015 e il 2016 e vede la collaborazione di tutte le Istituzioni pubbliche preposte al controllo delle Malattie Trasmesse da Alimenti (MTA).
Il coordinamento di tutte le attività e delle diverse competenze ha consentito di gestire un elevato numero di ceppi batterici con tempistiche ottimali ed una buona ripartizione del lavoro.
La collaborazione con l'LNR ha permesso di acquisire competenze tecnico scientifiche che si sono concretizzate nell'acquisizione di nuovi protocolli diagnostici per la caratterizzazione di Lm quali la determinazione del sierogruppo mediante Multiplex PCR.
Pur non essendo tra le tecniche di caratterizzazione con più elevato potere discriminante, la determinazione del sierogruppo/sierotipo dei ceppi di Lm ha consentito di stabilire un nesso tra i ceppi e le tipologie di alimenti stimandone il ruolo nelle infezioni umane (attribuzione delle fonti).
L'elevata frequenza di riscontro del sierotipo 1/2c, sia sul complesso dei ceppi analizzati che a livello di alimenti, potrebbe essere riconducibile all'elevato numero di salumi analizzati e dei relativi isolati dei campioni positivi. Il sierotipo 1/2a, risultato il secondo più frequente, oltre ad essere molto rappresentato tra gli isolati alimentari lo è stato anche tra quelli ambientali. Il sierotipo1/2a è uno di quelli più comunemente associati a casi umani di listeriosi (Montero et al., 2015) e la sua circolazione nel territorio, negli alimenti e negli stabilimenti di produzione e trasformazione, rappresenta un potenziale rischio per la salute pubblica. Anche il sierotipo 4b è fra quelli più di frequente isolati da casi clinici e si è rivelato spesso responsabile di focolai epidemici. Come nello studio condotto a livello nazionale da Gianfranceschi et al. (2009), questo sierotipo è stato rinvenuto, sebbene in maniera poco diffusa, anche a livello di stabilimenti di lavorazione e di alimenti pur non essendo comune in queste matrici.
La tipizzazione molecolare mediante PFGE ha evidenziato, nel territorio oggetto del lavoro, la circolazione di una popolazione batterica piuttosto eterogenea, dato questo che andrebbe approfondito con tecniche genomiche di nuova generazione e con più elevato potere discriminante quali il NGS.
La scelta attuata in laboratorio di isolare e caratterizzare fino a 5 ceppi per ciascun campione positivo ha trovato conferma nell'evidenza della presenza di 4 cloni differenti di Lm nello stesso alimento e allo stesso tempo ha confermato la necessità di utilizzare tecniche analitiche ancora più discriminanti quali il sequenziamento dell'intero genoma.
L'utilizzo di più tecniche di caratterizzazione con diverso potere discriminante si rivela un approccio vincente non solo per individuare la fonte di infezione nell'ambito di casi di listeriosi, ma anche più in generale per studiare la popolazione microbica associata ad un determinato territorio. Nel contesto della sorveglianza epidemiologica, infatti, i metodi di tipizzazione molecolare possono essere usati per monitorare la diffusione geografica e i cambiamenti prevalenti di cloni epidemici o endemici con l'obiettivo di una valutazione a lungo termine di strategie preventive o per il controllo di infezioni emergenti e riemergenti.
Ringraziamenti:
Si ringrazia tutto il personale tecnico dei laboratori che ha supportato l'isolamento, la collezione e la caratterizzazione dei ceppi di L. monocytogenes nell'anno in cui è stato svolto del lavoro.
Bibliografia
D'Agostino M., Wagner W., Vazquez-Boland J.A., Kuchta T., Karpiskova R., Hoorfar J., Novella S., Scortti M., Ellison J., Murray A., Fernandes I., Kuhn M., Pazlarova J., Heuvelink A., Cook N. (2004). A Validated PCR-Based Method To Detect Listeria monocytogenes Using Raw Milk as a Food Model - Towards an International Standard. Journal of Food Protection 67(8): 1646-1655.
Decisione della Commissione del 28 aprile 2008 recante modifica della decisione 2002/253/CE che stabilisce la definizione dei casi ai fini della dichiarazione delle malattie trasmissibili alla rete di sorveglianza comunitaria istituita ai sensi della decisione n. 2119/98/CE del Parlamento europeo e del Consiglio.
Decisione n. 1082/2013/UE del Parlamento Europeo e del Consiglio del 22 ottobre 2013 relativa alle gravi minacce per la salute a carattere transfrontaliero e che abroga la decisione n. 2119/98/CE
Di Pinto A., Novello L., Montemurro F., Bonerba E., Tantillo G. (2010). Occurrence of Listeria monocytogenes in ready-to-eat foods from supermarkets in Southern Italy. New Microbiologica, 33: 249-252.
Doumith M., Buchrieser C., Glaser P., Jacquet C., Martin P. (2004). Differentiation of The Major Listeria monocytogenes Serovars by Multiplex PCR. Journal of Clinical Microbiology 42(8):3819-3822.
EC (European Community). 2005. Commission regulation (EC) No. 2073/2005. Microbiological criteria for foodstuffs. Official Journal, 338, 1, 22.12.2005.
European Food Safety Authority (EFSA), (2017). The European Union summary report on trends and sources of zoonoses, zoonotic agents and food-borne outbreaks in 2016. Efsa Journal 15(12):5077 doi: 10.2903/j.efsa.2017.5077.
Gianfranceschi M.V., D'Ottavio M.C., Gattuso A., Bella A., Aureli P., (2009). Distribution of serotypes and pulsotypes of Listeria monocytogenes from human, food and environmental isolates (Italy 2002-2005). Food Microbiology 26: 520-526.
Hyden P., Pietzka A., Lennkh A., Murer A., Springer B., Blaschitz M., Indra A., Huhulescu S., Allenberger F., Ruppitsch W., Sensen C.W., (2016). Whole genome sequence-based serogrouping of Listeria monocytogenes isolates. Journal of Biotechnology 235: 181-186.
Montero D., Bodero M., Riveros G., Lapierre L., Gaggero A., Vidal R. M., Vidal M., (2015). Molecular epidemiology and genetic diversity of Listeria monocytogenes isolates from a wide variety of ready-to-eat foods and their relationship to clinical strains from listeriosis outbreaks in Chile. Frontiers in microbiology 6: 1-8. doi: 10.3389/fmicb.2015.00384
Pettengill J.B., Luo Y., Davis S., Chen Y., Gonzalez-Escalona N., Ottesen A., Rand H., Allard M.W., Strain E., (2014). An evaluation of alternative methods for constructing phylogenies from whole genome sequence data: a case study with Salmonella. 2: e620; DOI 10.7717/peerj.620.
Thomas E.J., King R.K., Burchak J., Gannon V P. (1191). Sensitive and Specific detection of Listeria monocytogenes in milk and ground beef with the polymerase chain reaction. Applied and Environmental Microbiology 57(9): 2576-2580.

Guidi et al, 2018 - Sorveglianza molecolare sui ceppi di
Listeria monocytogenes isolati nell’Umbria e nelle Marche nel 2017 (SPVet.it 108/2018)




 border="0">
border="0">








