Biblioteca Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche
Sanità Pubblica Veterinaria: Numero 100, Febbraio 2017 [http://www.spvet.it/] ISSN 1592-1581
Documento reperibile all'indirizzo: http://spvet.it/indice-spv.html#653
Aspetti sanitari della popolazione urbana di Columba livia in Umbria
Public health aspects of the urban population of Columba livia in Umbria (Italy)
Valeria Tammaro1, Ludovico Dipineto2, Paola Papa1, Livia Moscati1
1 Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche. Perugia
2 Università di Napoli Federico II - Dipartimento di Medicina Veterinaria e produzioni animali
Estratto della Tesi finale
Scuola di Specializzazione in Tecnologia e Patologia delle specie Avicole, del Coniglio e della Selvaggina
Dipartimento di Medicina Veterinaria e Produzioni Animali.
Università degli Studi di Napoli Federico II.
Anno Accademico 2015-2016.
Abstract. The study covers the monitoring activities, from March to July 2016, in order to assess health conditions of pigeons population and zoonotic risk in the urban environment, as well as organize activities aimed at limiting this Synanthropic species. These data were compared with those provided by IZS UM (Italy) "Osservatorio Epidemiologico Veterinario - Epidemiological Veterinary Observatory" already published. The tests show that the trend of Salmonella shows a fluctuating situation. Campylobacter coli, Campylobacter jejuni and Salmonella sp, where not found positive in the period, While Campylobacter jejuni shows an analogy with what we have observed for Salmonella.
Chlamydia psittaci shows a similar situation to Campylobacter jejuni.
The zoonotic potential of this pathogen is confirmed by PCR positivity, observed in the 2016 work.
Although the presence of Clamydia and Campylobacter jejuni significantly higher than Salmonella and Campylobacter coli, their common decline from 2007 seems to indicate the urgent need of preventative actions to prevent possible zoonoses.
From the very beginning, the parasites, especially Trichomonas and coccidi, are higher than tapeworms (Taenia sp.). Their trend shows a strong increase around 2011, followed by a continuous decline until nowadays, while ascariasis shows to be costant.
The surveys carried out from 2003 to 2016 show that proper management of Columba livia in Umbria has reduced the risk of zoonotic diseases transmission.
Bacterial and parasitic health hazards are mainly due to the large population of pigeons and the consequent high fecal contamination of the urban environment.
Removing the feces is of a primary importance in order to prevent (through vacuum cleaners and fans) the dirt powders enter in apartments, restaurants, offices, hospitals and schools, infecting the environment.
The present study also indicates that the urban pigeon may be taken into consideration not only as a possible vector of zoonoses, but also as a strategic bioindicator of environmental pollution
Riassunto. Lo studio riguarda le attività di monitoraggio condotte, in vista di azioni finalizzate al contenimento della popolazione di piccioni urbani, effettuate da Marzo a Luglio del 2016, al fine di valutarne le condizioni sanitarie ed il rischio zoonosico che la loro presenza comporta. Tali dati sono stati comparati con quelli forniti dall' Osservatorio Epidemiologico Veterinario dell'Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche già oggetto di pubblicazioni. Le prove evidenziano come l'andamento della Salmonella mostra una situazione altalenante, mentre per quanto riguarda il Campylobacter coli non si rilevano positività nel periodo, mentre il Campylobacter jejuni mostra un'analogia con quanto abbiamo osservato per la Salmonella.
Clamydia psittaci mostra una situazione analoga al Campylobacter jejuni. Il potenziale zoonosico di tale patogeno viene confermato dalla positività in PCR osservata nel lavoro del 2016. Pur essendo la presenza di Clamydia e Campylobacter jejuni significativamente maggiore rispetto a Salmonella e Campylobacter coli, il loro comune declino dal 2007 ad oggi, sembra indicare la non urgenza di mettere in campo azioni preventive per evitare possibili zoonosi da esse derivanti. Sin dall'inizio la presenza dei parassiti, soprattutto Trichomonas e coccidi, risulta essere maggiore rispetto alle tenie e che il loro andamento veda un forte incremento intorno al 2011, con un rapido declino fino ai giorni nostri, mentre la presenza di Ascaridi si mantiene costante. Le indagini effettuate dal 2003 al 2016 mostrano come una corretta gestione della popolazione di Columba livia in Umbria, abbia ridotto il rischio di trasmissione di malattie a carattere zoonotico.
I rischi sanitari batterici e parassitari, sono derivanti prevalentemente dalla elevata popolazione di piccioni e conseguentemente dall'alta contaminazione fecale dell'ambiente urbano. La rimozione di queste è un aspetto di primaria importanza per evitare che, tramite l'uso di aspiratori e ventilatori, polveri di deiezioni secche possano entrare negli appartamenti, ristoranti, uffici, ospedali, scuole infettando gli ambienti. Il presente studio indica anche che il piccione urbano potrebbe essere analizzato non solo come possibile vettore di zoonosi, ma anche come bioindicatore di inquinamento ambientale
Introduzione
Il Piccione di città (Columba livia forma domestica), è un organismo sinantropo che si rileva nelle aree abitate in gruppi numericamente estesi. Lo sviluppo nei centri urbani è dovuto ad uno stretto rapporto di commensalismo con l'uomo (Mancini 2002).
Attualmente i piccioni di città sono diffusi in tutto il mondo e costituiscono un rischio per la salute pubblica (Jerolmack 2008). L'incremento numerico delle popolazioni di questi animali desta preoccupazione in quanto tendono a colonizzare piazze, parchi pubblici imbrattando edifici e monumenti con gli escrementi e degradando manufatti pregevoli situati in città d'arte (Amadori et al., 2003).
Sul piano sanitario, ci sono poi i rischi per bambini, anziani e soggetti immunocompromessi che possono contagiarsi con deiezioni polverizzate, che si disperdono per via anemofila (Haag Wackermagel e Moch 2004).
In contesti come questi, i piccioni di città hanno importanza sul piano epidemiologico in quanto possono essere reservoir e potenziali vettori di un gran numero di malattie zoonotiche.
Alexander et al. (1985) e Haag-Wackernagel (2011), descrivono circa 110 agenti zoonotici isolati da piccioni. Gli enteropatogeni sono certamente uno degli agenti infettivi di maggiore attenzione (Gargiulo et al., 2014). Sebbene alcuni patogeni siano noti da tempo come Salmonella spp, Campylobacter spp, E. coli produttori di tossine, sembra che le zoonosi più diffuse da Columba livia nelle città siano Cryptococcus neoformans, Candida albicans e Chlamydophila psittaci (Horvatec Tomic et al., 2013).
Naturalmente la presenza di patogeni legati ai piccioni in determinate aeree non causa necessariamente patologie nell'uomo che sono legate anche ad altri fattori quali la via di trasmissione, la dose infettante, lo stato immunitario del soggetto.
Questione sanitaria e managment
L'amministrazione comunale di Perugia ha tentato nel tempo di contenere la popolazione di piccione domestico localizzata nel centro storico di città. Una serie di studi condotti a partire dal 1990 hanno consentito di avere informazioni sullo stato sanitario degli animali, sulla consistenza numerica, sulla densità della popolazione, sul numero di coppie nidificanti, sulla densità dei nidi, sulla localizzazione dei principali "dormitori" e dei luoghi di nidificazione.
Sono stati successivamente eseguiti vari interventi diretti, finalizzati al controllo numerico della popolazione, ai quali sono seguiti ulteriori censimenti per valutarne l'efficacia.
Una delle prime indagini sanitarie sulle popolazioni di piccione urbano effettuata in Umbria è riportata da Montefameglio et al (2003). Dalle analisi svolte per questo lavoro si sono registrate delle positività per la ricerca di Campylobacter termofili e di anticorpi per Toxoplasma gondii e Chlamydophila psittaci.
Attraverso un'indagine condotta dall'Istituto Zooprofilattico dell'Umbria e delle Marche da giugno 2004 a dicembre 2006 su 142 animali esaminati è stata accertata la presenza di Salmonella Typhimurium e di Campylobacter termofili (Grelloni et al., 2007).
Nello stesso lavoro sono stati ritrovati anticorpi nei confronti di Toxoplasma gondii e Chlamydophila psittaci.
Nel periodo che va dal mese di Marzo 2010 al mese di Gennaio 2011 (Maresca et al., 2011), sono stati esaminati 75 piccioni catturati nelle attività di bonifica cittadina, riscontrando positività per Salmonella spp, Campylobacter termofili e anticorpi nei confronti di Toxoplasma gondii e Chlamydophila psittaci.
Tutti e tre i lavori sopradescritti hanno riscontrato la positività di molti animali ai più diffusi endoparassiti (tenie, coccidi, ascaridi, capillarie)

Figura 1. Il piccioni fanno ormai parte dell'ambiente urbano in ogni luogo, ben adattati alla vicinanza con l'Uomo (Chiostro di Santa Chiara, Napoli)
Figure 1. Pigeons are now part of the urban environment everywhere, Well adapted to the proximity of Man (St. Chiara Cloister, Naples - Italy)
Foto V. Tammaro (2016). Licenza Creative Commons. Attribution-NonCommercial 4.0 International [CCBYNC4.0]
Scopo del lavoro
Il presente lavoro ha avuto lo scopo di valutare lo stato sanitario della popolazione di Columba livia stanziale in un'area urbana della città di Perugia, in relazione al potenziale rischio per l'Uomo, gli animali domestici e selvatici. Ciò anche al fine di aggiornare le conoscenze relative ai patogeni trasmessi da tale specie sinantropa.
In questo studio, oltre a controllare lo stato di diffusione di malattie già oggetto di indagini precedenti, è stata analizzata la presenza di patogeni emergenti come Giardia duodenalis, Cryptococcus neoformans, Coxiella burnetii.
Materiali e Metodi
Il lavoro su Columba livia è basato sul set di dati sperimentali (forniti dal laboratorio di Diagnostica dell'IZS-UM) relativi ad un campionamento sulla popolazione di piccioni in località di Sant'Andrea delle Fratte.
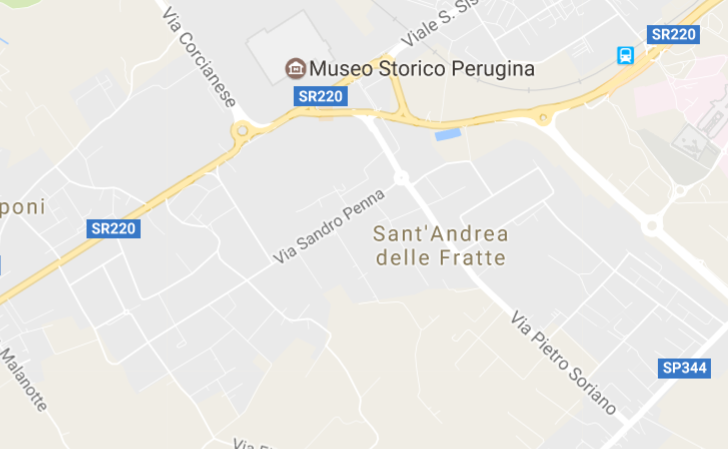
Figura 1: Area di studio e campionamento, località Sant'Andrea delle Fratte, Perugia.
Figure 1: Study and sampling area, "Sant'Andrea delle Fratte", Perugia, Italy
Le catture dei volatili sono state effettuate nel periodo da marzo a luglio dell'anno 2016.
Ai campioni è stato assegnato dal Servizio Accettazione dell'IZSUM un numero identificativo che accompagna il campione stesso e tutti i sub-campioni che da esso possono essere generati.
I laboratori dell'IZSUM si avvalgono di un sistema informatico di gestione dei campioni denominato S.I.G.L.A. che permette di identificarli, di trasferirli, di inserire le risposte e di spedirle per via telematica.
Le determinazioni previste per il controllo sanitario della popolazione di volatili sono state le seguenti:
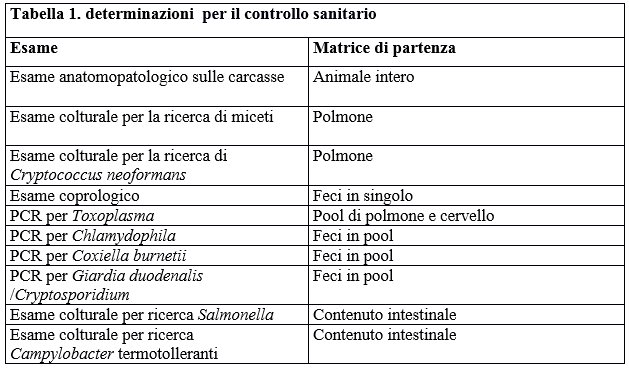
Table 1. Determinations for health control
La procedure di seguito descritte sono quelle in uso presso il Laboratorio di Diagnostica dell'Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche (sede centrale di Perugia).

Figura 2 - a. Campioni sul tavolo anatomico
Figure 2 - a. Sample on the anatomical table
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)
ESAME ANATOMOPATOLOGICO
L'esame anatomopatologico è stato effettuato sui volatili seguendo le tecniche descritte nel libro: "tecnica autoptica e diagnostica cadaverica" (Taccini et al., 2006).
Sono state effettuate in ordine le seguenti valutazioni:
- Esame visivo esterno del piumaggio
- Esame della cute
- Esame delle mucose apparenti (in particolare quella orale e oculo-congiuntivale)
- Esame dello scheletro e delle articolazioni
- Esame della testa (alterazioni di volume mono o bilaterali)
- Esame delle ali e delle zampe
- Apertura del becco e del collo (per escludere la presenza di membrane, pseudomembrane, granulomi)
- Scuoiamento con esame del sottocute (eventuali edemi o emorragie)
- Esame delle masse muscolari
- Apertura della cavità splancnica con osservazione degli organi toraco-addominali e del peritoneo
- Esame dei sacchi aerei (eventuale essudato o granulomi).
- Esame del fegato (eventuali alterazioni di colore, volume o consistenza e eventuali lesioni macroscopiche)
- Esame dell'apparato digerente (proventriglio, ventriglio, intestino tenue e grosso con particolare attenzione ai ciechi)
- Esame della milza (eventuali alterazioni di colore, volume o consistenza e eventuali lesioni macroscopiche)
- Esame dell'apparato riproduttore maschile e femminile, borsa di Fabrizio e cloaca
- Esame dell'apparato respiratorio a partire dai seni infraorbitali, sacchi aerei e polmoni
- Esame dell'apparato urinario (eventuali depositi di cristalli sulla superficie renale)
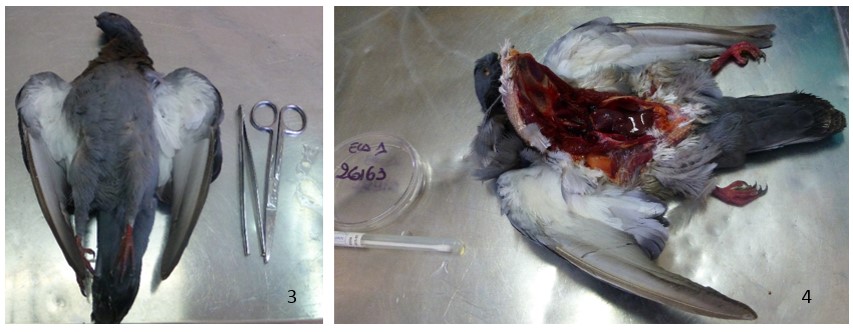
Figura 3. Posizione del capo morto ed esame esterno. Figura 4. Ribaltamento dello sterno dopo la resezione delle coste
Figure 3. Position of pigeons and examination. Figure 4. overturning the sternum after the rib resection
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)
La procedura prevede che qualora si ravvisino alterazioni o lesioni, l'anatomopatologo possa richiedere ulteriori esami (batteriologico, virologico, istologico, parassitologico) per poter avere ulteriori informazioni da utilizzare per la diagnosi.
ESAMI COLTURALI
Esame Micologico
L'esame, eseguito seguendo la procedura standard in uso presso il laboratorio di Diagnostica dell'IZSUM (Polinelli et al., 1994; Andreoni et al., 2003; Vidotto 2002), permette l'identificazione di muffe non dermatofitiche e lieviti ad eccezione dei generi Cryptococcus e Malassezia, per i quali sono previste specifiche procedure.
Per ogni campione di polmone è stata contrassegnata una piastra di Saboraud Dextrose Agar (SDA) con il numero di accettazione e la data.
Dopo aver sterilizzato la superficie dell'organo con una spatola riscaldata alla fiamma di un becco Bunsen, viene isolato un frammento dell'organo utilizzando pinze e forbici sterili.
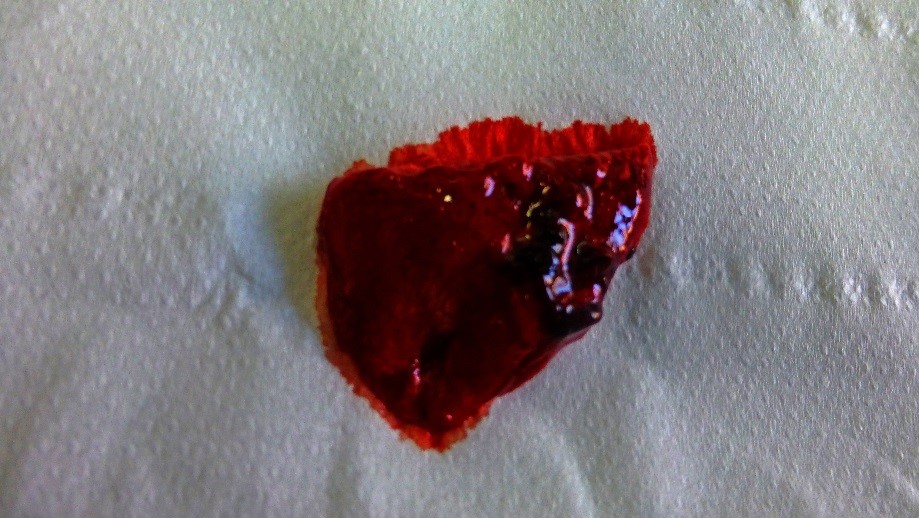
Figura 5. Esame del polmone
Figure 5. Lung examination
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)

Figura 6. Flambatura della superficie dell'organo
Figure 6. Burning of the organ surface
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)
Figura 7. Inserimento del tampone
Figure 7. Inserting the swab
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)

Figura 8. Piastra contrassegnata SDA
Figure 8. SDA labeled plate
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)
Semina effettuata per diluizione:
La parte interna del frammento di polmone prelevato va strisciata sulla superficie della piastra di SDA dividendola idealmente in quattro quadranti ed eseguendo la strisciata solo su un quadrante.
Sterilizzare quindi l'ansa e partire da un punto della superficie del primo quadrante seminato, prolungando con la stessa la semina degli altri quadranti, avendo cura di flambare e raffreddare l'ansa tra un quadrante e l'altro.
Incubare le piastre capovolte a 37°C ± 1°C in atmosfera normale per 24-48-72 ore. La prima lettura delle piastre va eseguita dopo 24 ore di incubazione. Qualora dopo 24 ore non vi sia sviluppo fungino o questo sia molto ridotto, prolungare l'incubazione per ulteriori 24-48 ore.
Alla lettura delle piastre vanno isolate le colonie che abbiano un aspetto riferibile a muffe non dermatofitiche (micelio cotonoso o vellutato o fioccoso, di colore e profilo variabile) o di lieviti (patina cremosa di colore e odore variabili e di consistenza simile a quella di una colonia batterica).
Passaggio delle colonie:
A partire da ogni singola colonia sospetta vanno allestite delle subcolture su SDA con la tecnica della semina per diluizione.
Incubare le piastre capovolte a 37°C ± 1°C in atmosfera normale per 24-48-72 ore.
|
Identificazione delle muffe non dermatofitiche
Per l'identificazione delle muffe non dermatofitiche bisogna considerare le caratteristiche macro e microscopiche delle stesse.
Dal punto di vista macroscopico vengono valutati i seguenti parametri: tempo di crescita della colonia, diametro, margini (netti, regolari, frastagliati, etc...), tessitura (cotonosa, fioccosa, granulosa, etc ...), colore sia della superficie (recto) che del fondo (verso).
Dal punto di vista microscopico (eseguito con la tecnica descritta di seguito) vengono valutati, se presenti, i seguenti elementi: ife (presenza o meno di setti, presenza o meno di ramificazioni, tipologia di ramificazione), strutture riproduttive quali spore e conidi (morfologia, presenza o meno di setti, aspetto della parete (regolare, frastagliata, echinulata etc…), conidiofori (ife specializzate su cui generano le cellule conidiogene), sporangi (strutture a forma di sacco, racchiudenti spore).
Esame microscopico a fresco delle colonie fungine:
Porre una goccia di blu di metilene su un vetrino porta-oggetto.
Asportare con nastro adesivo un frammento di colonia e adagiare il nastro adesivo sul vetrino porta-oggetto col blu di metilene.
Far aderire bene il nastro adesivo alla superficie del vetrino.
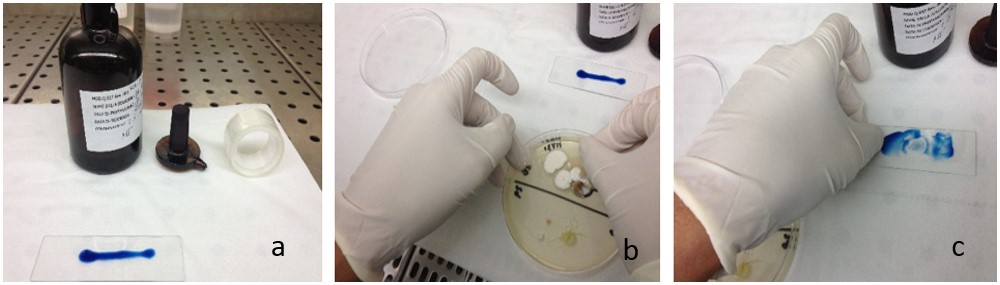
Figura 9 a, b, c. Fasi della preparazione del campione per l'osservazione al microscopio
Figure 9 a, b, c. Phases of sample preparation for microscopic observation
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)
Osservare il vetrino al microscopio a basso (40X) e alto ingrandimento ad immersione (100X).
Riportare sul foglio di lavoro la descrizione microscopica della colonia unitamente alle informazioni già registrate sull'aspetto macroscopico.
Nel caso in cui l'esito dell'identificazione macro e microscopica non dia un risultato soddisfacente si può decidere di procedere ad una diagnosi molecolare, attraverso le seguenti fasi:
- estrazione del DNA;
- amplificazione del DNA PCR end- point con i Primer
- ITS4 (5'-TCC TCC GCT TTA TTG ATA TGC -3')
- ITS5 (5'-GGA AGT AAA AGT CGT AAC AAG G -3')
- elettroforesi in gel di Agarosio;
- sequenziamento genico.
Figura 10. Eletroforesi del DNA in gel di agarosio
Figure 10. DNA elytrophoresis in agarose gel
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)
|
Identificazione dei lieviti
Accanto ad una valutazione macroscopica delle colonie ottenute in purezza, di cui si valutano il tempo di crescita (in ore), il diametro, i margini (netti, regolari, frastagliati, etc...) e l'aspetto (cremoso, rugoso, etc…), si procede all'identificazione di genere e/o specie utilizzando i sistemi miniaturizzati in commercio. Il laboratorio attualmente utilizza il Kit ID32C della Ditta Biomerieux® secondo le istruzioni indicate dal produttore.
Nel caso in cui l'esito dell'identificazione in micrometodo non dia un risultato soddisfacente si può decidere di procedere ad una diagnosi molecolare, attraverso le seguenti fasi:
- estrazione del DNA;
- amplificazione del DNA PCR end- point con i Primer
- NL1 (5'-GCATATCAATAAGCGGAGGAAAAG-3')
- NL4 (5'-GGTCCGTGTTTCAAGACGG-3')
- elettroforesi in gel di Agarosio;
- sequenziamento genico.
Cryptococcus spp - esame colturale:
L'esame, eseguito seguendo la letteratura accreditata (Polonelli et al., 1994; Andreoni et al., 2003; Vidotto 2002) e la procedura standard in uso presso il laboratorio di Diagnostica dell'IZSUM (DV PRT.LDPG1.001 rev.000 "Dossier di validazione Isolamento ed identificazione Cryptococcus spp), permette l'isolamento e l'identificazione di lieviti appartenenti al genere Cryptococcus.
Per ogni campione di polmone è stata contrassegnata una piastra di CAFC (Caffeic Acid Ferric Citrate) medium con il n° di accettazione e la data.
Dopo aver sterilizzato la superficie dell'organo con una spatola riscaldata alla fiamma di un becco Bunsen, viene isolato un frammento dell'organo con pinze e forbici sterili.
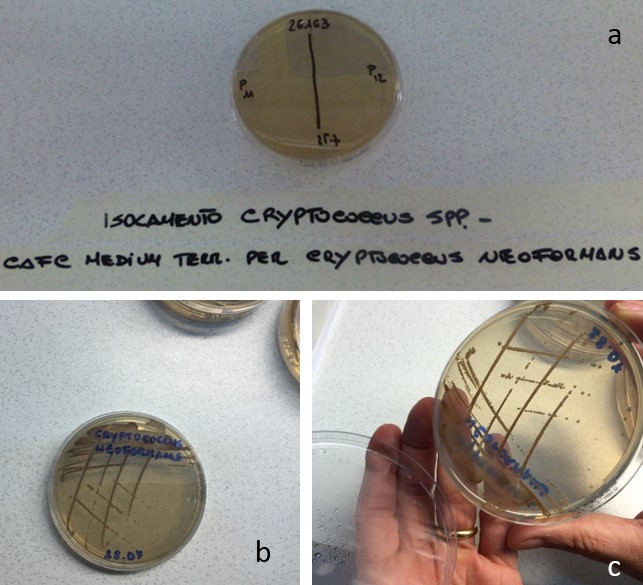
Figura 11 a, b, c. Isolamento Cryptococcus spp su piastra
Figure 11 a, b, c. Cryptococcus sp isolation on plate
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)
Semina effettuata per diluizione:
La parte interna del frammento di polmone prelevato va strisciata sulla superficie della piastra di SDA dividendola idealmente in quattro quadranti ed eseguendo la strisciata solo su un quadrante. Sterilizzare quindi l'ansa e partire da un punto della superficie del primo quadrante seminato, prolungando con la stessa la semina degli altri quadranti, avendo cura di flambare e raffreddare l'ansa tra un quadrante e l'altro.
Incubare le piastre capovolte a 25°C ± 1°C in atmosfera normale per 48-72 ore. Viene eseguita la prima lettura delle piastre dopo 48 ± 3 ore di incubazione; qualora, dopo le 48 ore, non vi sia sviluppo fungino o questo sia molto ridotto, l'incubazione viene prolungata per altre 24 ± 3 ore .
In caso di crescita di colonie riferibili a lievito, ovvero con un aspetto cremoso o gelatinoso, lisce o convesse e di colore variabile dal bianco al marrone, si esegue una subcoltura.
Passaggio delle colonie da sottoporre ad identificazione:
Partendo dalla singola colonia allestire una subcoltura su CAFC, con la tecnica della semina per diluizione; incubare la subcoltura capovolta a 25°C ±1°C in atmosfera normale per 48±3h, quindi eseguire sulla colonia singola una colorazione di Gram.
Nel caso in cui la colonia risulti Gram + e di morfologia riconducibile a lievito, prolungare l'incubazione fino a 5 + 2gg, in modo che nel caso sia C. neoformans avrà aspetto liscio, convesso, traslucente, gelatinoso o mucoso e pigmentazione di colore bruno-nero, grazie all'attività fenolo-ossidasica del lievito; in caso di lievito diverso da C. neoformans, si avrà lo stesso aspetto macroscopico ma con colore variabile dal crema al beige.
|
ESAMI PARASSITOLOGICI
Gli esami parassitologici sono stati effettuati seguendo le procedure standard in uso presso il laboratorio di parassitologia dell'Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche. I particolari delle metodiche applicate, sono riportati di seguito.
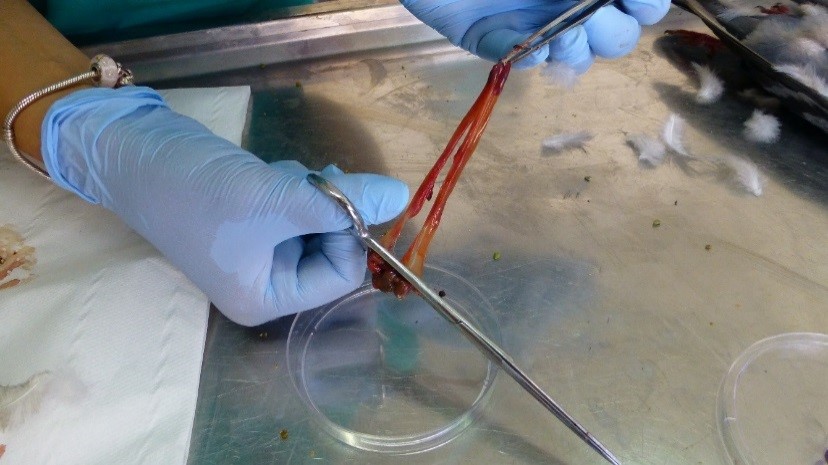
Figura 12. Prelievo del contenuto dei ciechi
Figure 12. Taking the cecum content
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)
Esame copromicroscopico qualitativo:
L'esame effettuato, seguendo la procedura standard in uso presso il laboratorio di Diagnostica dell'IZSUM (Bpch e Superer 1980; Ambrosi 1995) permette l'individuazione di uova di elminti e di oocisti di coccidi (protozoi).
La tecnica dell'esame coprologico, si effettua a partire da materiale fecale e permette di evidenziare oocisti, larve, larve di elminti e altre forme parassitarie che sono presenti nel frammento esaminato.
Arricchimento per flottazione:
L'esame prevede di sospendere il campione di feci in una soluzione il cui peso specifico sia maggiore di quello delle uova/oocisti ed inferiore a quello delle scorie fecali. In tal modo le forme parassitarie (uova ed oocisti), flottando, si concentrano alla superficie del liquido e possono essere raccolte ed identificate morfologicamente. Le uova di cestodi e nematodi flottano in soluzioni con un peso specifico fra 1.100-1.200.
Preparazione del campione:
Una quantità di circa 1 gr di feci (prelevato a livello di intestino tenue e dei ciechi) viene sminuzzata in un mortaio mescolando gradatamente con 15 ml di soluzione di flottazione (cloruro di sodio densità = 1.200) sino ad ottenere una sospensione il più omogenea possibile. Filtrare circa 10 ml di sospensione attraverso uno strato di garza posto in un imbuto infilato in una provetta di polipropilene identificata con il numero di accettazione, al fine di trattenere il materiale più grossolano. Si aggiunge la soluzione di flottazione mediante pipetta pasteur monouso fino a formare un menisco all'apice della provetta. Sul menisco si appoggia delicatamente un vetrino copri-oggetto poco più grande del diametro della provetta che si lascia in situ per 20 minuti. Trascorsi i venti minuti, il copri-oggetto viene trasferito su un vetrino portaoggetti per l'osservazione avendo cura, durante il sollevamento ed il trasferimento, di mantenerlo perfettamente orizzontale.
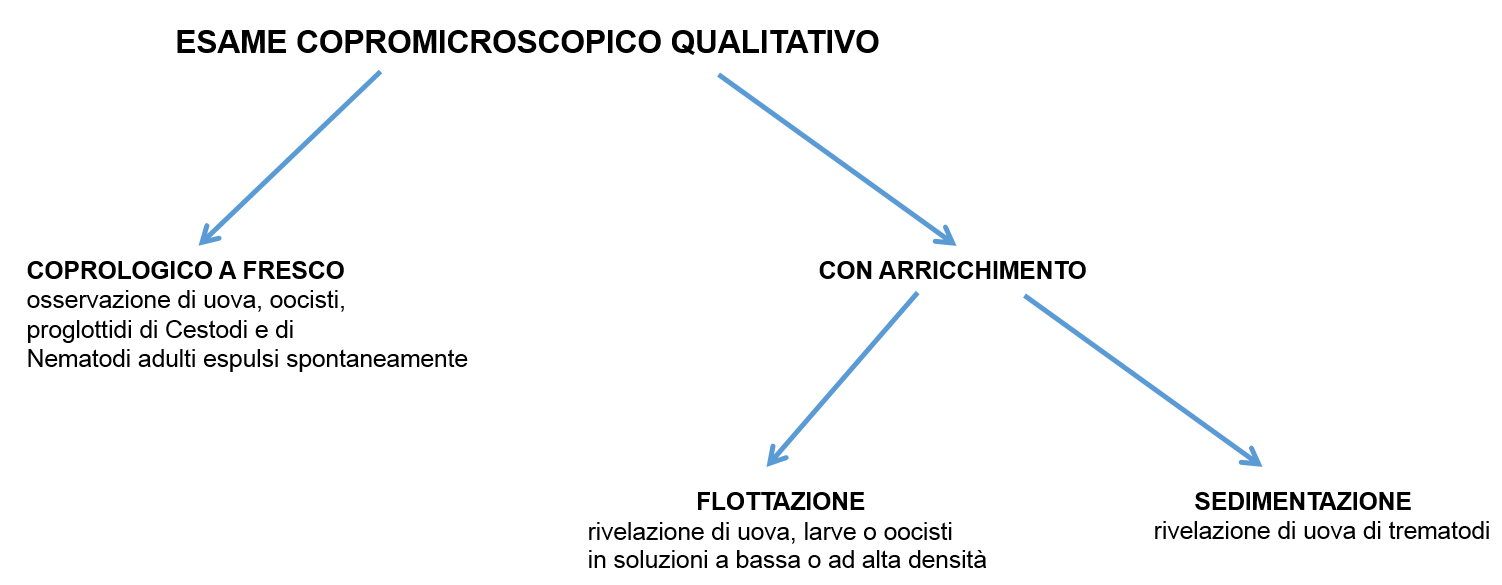
Figura 13. Rappresentazione schematica del procedimento
Figure 13. Schematic representation of the procedure
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)

Figura 14. Preparazione campione coprologico
Figure 14. Coprological sample preparation
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)
Lettura del vetrino:
I preparati sono stati osservati al microscopio ottico all'ingrandimento di 10 X 10 e/o se necessario di 10 X 20 avendo cura di esplorare tutta la superficie del vetrino copri oggetto.
Identificare le uova e/o oocisti osservate seguendo lo schema interpretativo consigliato dal testo "Parassitologia e Malattie Parassitarie degli animali" citato in bibliografia e qui brevemente riassunto:
Strongili gastrointestinali: uova di forma ellittica, parete sottile, interno trasparente, morula centrale scura, numero variabile di blastomeri (numerosi in alcuni trichostrongilidi), diametro maggiore ± 75/80 µm.
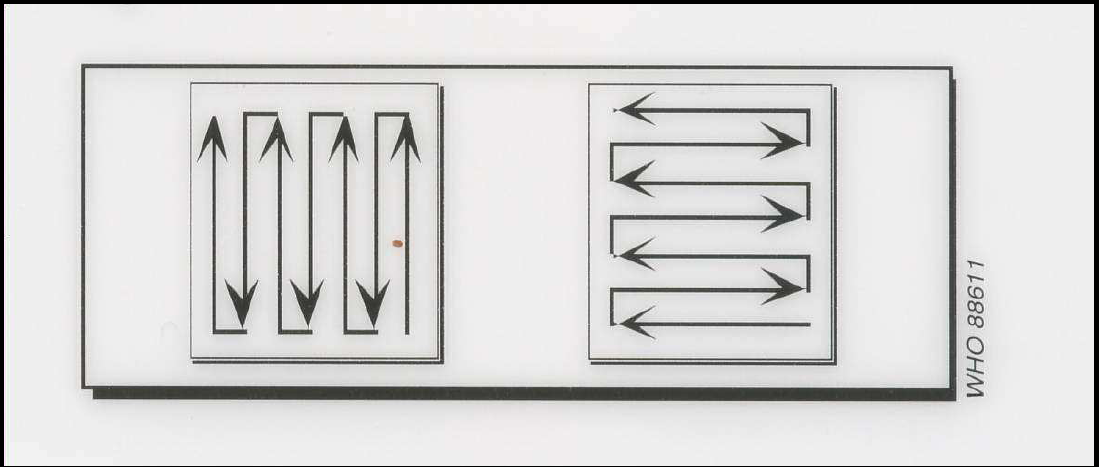
Figura 15. Lettura del vetrino microscopico: spostamenti verticali e laterali del tavolinetto traslatore
Figure 15. Reading the microscopic slide: vertical and lateral displacements of the translator table
: oocisti di forma rotonda o ellissoidale, con parete liscia sottile ed incolore, sporulate o asporulate.
Trichomonas spp: protozoo flagellato piriforme di circa 12-20 µm x 8-12 µm con tre-cinque flagelli e membrana ondulata.
Histomonas spp: protozoo flagellato pleomorfico ameboide di diametro variabile fra i 6-20 µm
Capillaria spp: uova a forma di barile, chiare, misurano 48-53 µm x 24 µm, hanno la parete spessa, lievemente striata e sono munite di tappi ai due poli.
Syngamus spp: uova ellissoidali a parete sottile, misurano 70-100 µm x 43-46 µm ed hanno uno spesso opercolo ad entrambe le estremità.
Ascaridia spp/ Heterakis spp: uova ovali con un guscio liscio e non facilmente distinguibile da quello di un altro comune nematode del pollame, Heterakis.
Raillietina tetragona: le uova presenti nelle capsule ovigere all'interno delle proglottidi misurano 65 µm x 90 µm
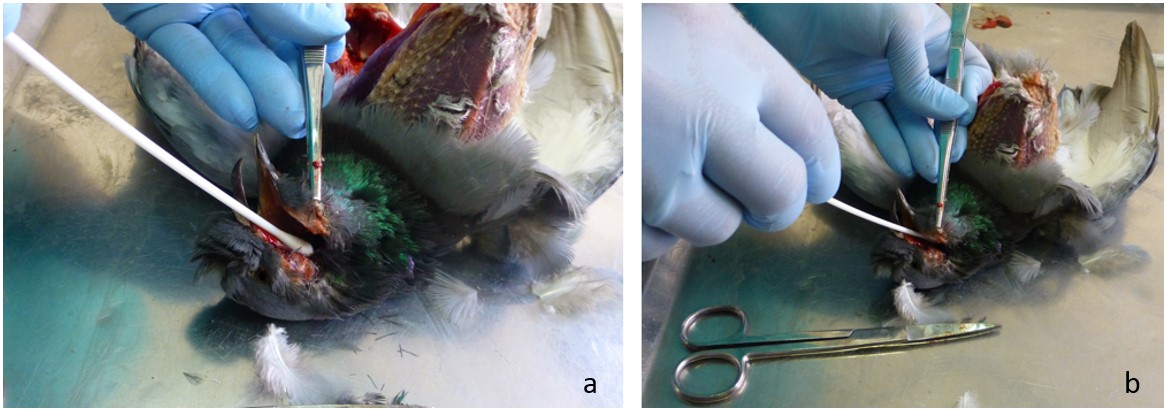
Figura 17 a, b. Apertura del becco e del collo (canale esofageo), per la ricerca del Trichomonas spp.
Figure 17a, b. Opening of the beak and neck (esophageal channel), for the search for Trichomonas
by IZSUM/2016 under Creative Commons Attribution 4.0 International License (CC BY 4.0)
|
ESAMI MOLECOLARI
Gli esami molecolari sono stati eseguiti su pool di organi o feci o singoli campioni seguendo le procedure in uso presso il laboratorio di Biologia Molecolare dell'Istituto Zooprofilattico Sperimentale dell'Umbria e delle Marche, di seguito riportate.
a. Estrazione del DNA;
b. Amplificazione del DNA (PCR);
c. Elettroforesi su gel di agarosio.
a) Estrazione del DNA
L'estrazione di DNA genomico ha seguito le metodiche accreditate (Boom et al., 1990; Vogelsteine Gillespie 1979) e le metodologie QIAamp DNA Mini Kit HandBook (QIAGEN)®, Protocol TaqMan® Sample to SNPTM Kit (DNA Extract All Lysis Reagents) ®
È stata eseguita a partire da omogenati mediante un kit commerciale che si basa sulla capacità del DNA e degli acidi nucleici in generale di adsorbirsi a particelle di silice in presenza di sali caotropici come Guanidina Idrocloruro (GuHCl) o Guanidina Isotiocianato (Gu SCN).
Gli ioni dei sali caotropici, carichi positivamente, creano un ponte cationico tra il DNA e la membrana di silice presente nella colonna, entrambi carichi negativamente.
Poiché in seguito alla lisi, gli acidi nucleici diventano facilmente aggredibili da parte delle proteasi cellulari (nucleasi), è necessario aggiungere un enzima (Proteinasi K) in grado di degradare le stesse e salvaguardare l'integrità degli acidi nucleici. L'aggiunta del tampone di eluizione a basso contenuto salino, permette il successivo distacco del DNA dalla Silice e la sua liberazione in soluzione.
b) PCR end-point
Il principio della PCR, (Kary Mullis 1986), sfrutta la capacità della DNA-polimerasi di sintetizzare altre molecole di acido nucleico sullo stampo di un campione dato, in modo da ottenere, in step successivi, l'amplificazione esponenziale del frammento stesso.
I componenti essenziali della PCR sono:
- 1. due inneschi oligonucleotidici di sintesi (primer) complementari a regioni dei filamenti opposti che fiancheggiano la sequenza di DNA bersaglio e che, dopo l'ibridazione con il DNA stampo abbiano le proprie estremità ossidriliche 3' orientate l'una verso l'altra;
- 2. una sequenza bersaglio nel campione di DNA che si collochi tra l'uno e l'altro innesco;
- 3. una DNA polimerasi termostabile capace di resistere a più di 95°C;
- 4. i quattro desossiribonucleotidi (Adenina, Guanina, Citosina, Timina);
- 5. MgCl2
Questi componenti miscelati in un opportuno buffer costituiscono la cosiddetta "mix" di reazione.
L'amplificazione consta di tre fasi fondamentali:
- denaturazione del DNA a 95°C in modo da provocare l'apertura dei due filamenti di acido nucleico;
- rinaturazione (50∼70°C) durante la quale avviene l'annealing (appaiamento) dei primer;
- allungamento: innesco della polimerasi con sintesi dei nuovi filamenti sullo stampo dei precedenti (∼ 72°C)
La reiterazione ciclica di queste fasi permette di accrescere esponenzialmente la quantità di partenza del DNA stampo.
La polimerasi in questione (Taq) è ottenuta dal batterio Thermus acquaticus, un microrganismo termofilo, ed è grazie alla sua caratteristica di termostabilità a temperature superiori a 95°C che è stata semplificata la reazione di amplificazione, altrimenti vincolata da macchinosi processi di ripristino dell'enzima.
I cicli di PCR possono essere semplificati utilizzando uno strumento automatico detto amplificatore o termociclatore. Tale strumento, una volta messa la "mix" nelle provette, con il relativo DNA, esegue l'intero processo di amplificazione senza ulteriore lavoro da parte dell'operatore.
Il DNA amplificato viene poi fatto correre in un gel di agarosio per la verifica dell'amplificazione.
c) Elettroforesi su gel
Metodo di separazione per macromolecole ed in particolare per proteine e frammenti di DNA; semplicità e velocità rendono tale sistema diffuso ed utilizzato.
Tale metodo di separazione è basato sulla diversa velocità di migrazione di particelle elettricamente cariche sotto l'influenza di un campo elettrico.
Il principio base è quello di un setaccio molecolare attraverso il quale le diverse molecole vengono fatte passare: la velocità di migrazione dipende dalla massa, dalla dimensione, dalla carica e dalla forma delle varie particelle, ossia dalla loro mobilità elettroforetica. Questa grandezza è il rapporto tra la velocità della particella (cm/s) e il campo elettrico utilizzato (Volt/cm).
La mobilità elettroforetica, essendo una funzione del rapporto tra carica e raggio, è diversa da una particella ad un'altra; applicando un campo elettrico ad una miscela ionica le varie specie migreranno con velocità diversa a seconda delle rispettiva mobilità. Frammenti di lunghezza minore sono meno ostacolati nella loro corsa attraverso le maglie del gel per cui si muovono a velocità maggiore rispetto a frammenti più lunghi. Alla fine di una corsa elettroforetica, quindi, frammenti più piccoli si troveranno nella parte bassa del gel mentre i frammenti più grossi saranno localizzati nella parte prossima al punto di caricamento
In condizioni standard la mobilità elettroforetica di molecole di DNA è proporzionale al logaritmo della loro massa. Per calcolare il peso molecolare dei frammenti viene effettuata una calibrazione con molecole di DNA di peso noto, detto marker.
Il DNA viene colorato con un "intercalante safe" (midori green-Nippon genetics Europe GmbH®), una molecola fluorescente alla luce ultravioletta, che si intercala nella doppia elica dell'acido nucleico.
La migrazione elettroforetica che avviene su un supporto solido di natura porosa imbevuto di una soluzione elettrolitica (tampone che permette il passaggio della corrente) prende il nome di elettroforesi zonale in quanto le sostanze restano separate in zone ben distinte.
|
Le tecniche molecolari sono state utilizzate per ricercare i seguenti patogeni:
- Coxiella burnetii;
- Chlamydia spp.;
- Toxoplasma gondii;
- Criptosporidium spp.;
- Giardia duodenalis.
PCR per Coxiella burnetii
L'esame, eseguito seguendo la procedura standard in uso presso il laboratorio di Diagnostica dell'IZSUM seguendo la letteratura (Berri et al., 2000; Magnino et al., 2009) viene effettuata su un pool di feci.
a) Estrazione del campione
Per l'estrazione del DNA genomico da feci il laboratorio ha utilizzato il kit QIAmp® Fast DNA Stool Mini Kit della Qiagen®.
- Preparare una serie di provette da 2 ml opportunamente identificate.
- Porre in ciascuna provetta un pool di 180-220 mg di feci e posizionare le provette su ghiaccio.
- Aggiungere 1 ml di InhibitEX Buffer a ciascun campione.
- Vortexare per 1 minuto fino ad omogenizzazione del materiale fecale.
- Riscaldare la sospensione per 5 minuti a 70°C.
- Vortexare per 15 secondi.
- Centrifugare il campione per 1 minuto.
- Preparare una nuova provetta da 2 ml correttamente identificata con 15 µl di Proteinasi K.
- Aggiungere alla provetta con proteinasi K 200 µl di surnatante della sospensione di feci.
- Aggiungere 200 µl di Buffer AL e vortexare per 15 secondi.
- Incubare a 70°C per 10 minuti.
- Aggiungere 200 µl di etanolo (96-100%) e vortexare.
- Prelevare delicatamente 600 µl del lisato e porlo nella colonnina QIamp spin.
- Centrifugare la colonnina per 1 minuto e cambiare il tubo di raccolta del filtrato.
- Aggiungere alla colonnina 500 µl di Buffer AW1.
- Centrifugare per 1 minuto.
- Cambiare il tubo di raccolta del filtrato.
- Aggiungere alla colonnina 500 µl di Buffer AW2.
- Centrifugare per 3 minuti.
- Cambiare il tubo di raccolta del filtrato.
- Centrifugare per 3 minuti.
- Trasferire la colonnina in una nuova provetta da 1.5 ml correttamente identificata e aggiungere 200 µl di Buffer ATE.
- Incubare per 1 minuto a temperatura ambiente e centrifugare per 1 minuto per eluire il DNA dalla colonnina.
- Richiudere le provette contenenti il DNA che, se non viene utilizzato subito, va conservato a -20°C.
La procedura in uso presso il laboratorio di Diagnostica dell'IZSUM è una PCR end point con profilo termico "touch-down".
I primi cinque cicli della reazione infatti, prevedono la diminuzione della temperatura di appaiamento (annealing) di un grado a ciclo. La strategia di reazione touch-down permette di rendere i primi cicli di PCR estremamente "stringenti", tali cioè da promuovere l'amplificazione solo di frammenti specifici rendendo instabili eventuali annealing dei primer a sequenze di DNA non perfettamente complementari.
In effetti una temperatura troppo bassa porta all'annealing dei primer a sequenze non esattamente complementari e quindi all'amplificazione di frammenti non specifici, mentre una temperatura troppo alta può ridurre la resa in quanto solo una frazione delle molecole del primer riesce ad innescare la polimerizzazione a causa dell'elevata instabilità del loro appaiamento con il DNA stampo.
Per ciascuna sessione di lavoro in cui vengono posti ad amplificare campioni in esame, vanno aggiunti un campione negativo di amplificazione (acqua bidistillata) e un campione positivo di amplificazione (DNA già estratto da un campione positivo già noto).
I primer utilizzati hanno le seguenti sequenze:
Primer COX F: 5'-TATGTATCCACCGTAGCCAGT-3'
Primer COX R: 5'-CCCAACAACACCTCCTTATTC-3'l
La mix della reazione è stata allestita utilizzando la GoTaq® DNA polimerasi e utilizzando le concentrazioni come di seguito indicato:
Il termociclatore è stato impostato per eseguire i cicli come da grafico:
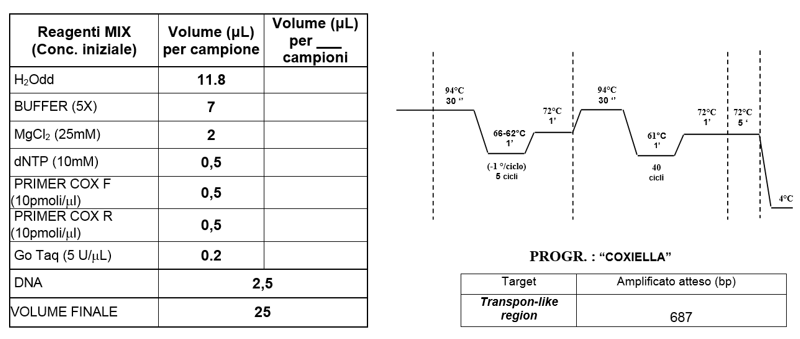
Figura 19. Mix di reazione e profilo termico di amplificazione
Figure 19. Reaction mix and thermal amplification profile
c) Interpretazione dei risultati
La lettura dei risultati, dopo corsa su gel di agarosio al 2%, viene effettuata verificando la presenza/assenza di amplificati, le cui dimensioni (687 bp) vengono confrontate con quelle dei controlli positivi di estrazione, di amplificazione e con quelle del marcatore di peso molecolare noto.
PCR -RFLP (Restriction Fragment of Lenght Polymorphism) per Chlamydia spp.
La procedura in uso presso il laboratorio di Diagnostica dell'IZSUM (Ossewaarde e Meijer 1999; Vicari et al., 2004) per l'identificazione della Chlamydia si basa sull'amplificazione mediante reazione a catena della polimerasi di una specifica sequenza bersaglio sul gene 16 S rRNA di Chlamydophila/Chlamydia; tale target essendo comune a diverse specie (psittaci, abortus, pecorum, trachomatis, felis, pneumoniae, suis) si presta ad una successiva analisi di restrizione enzimatica mediande endonucleasi di restrizione Tru 1 I o INF I, le quali producono enzimi di restrizione specifici che determinano il taglio del DNA o di una sua porzione a livello di sequenze enzima - specifiche originando una serie di frammenti di dimensione nota. L'analisi di tali frammenti consente la successiva tipizzazione delle specie Chlamydophila psittaci, Chlamydophila abortus, Chlamydophila pecorum, Chlamydophila felis, Chlamydophila pneumoniae, Chlamydophila trachomatis, Chlamydophila suis.
a) Estrazione del campione
Tale procedura si applica su DNA genomico estratto da pool di feci come precedentemente descritto.
La procedura in uso presso il laboratorio di Diagnostica dell'IZSUM è una PCR touch-down.
b) Amplificazione del DNA
I primi cinque cicli della reazione infatti prevedono la diminuzione di un grado a ciclo della temperatura di appaiamento (annealing).
Per ciascuna sessione di lavoro in cui vengono posti ad amplificare campioni in esame, vanno aggiunti un campione negativo di amplificazione (acqua bidistillata) e un campione positivo di amplificazione (DNA già estratto di un campione positivo già noto).
I primer utilizzati hanno le seguenti sequenze:
Primer CHLA 16S Forward : 5'-CGTGGATGAGGCATGCAAGTCGA-3'
Primer CHLA 16 S Reverse: 5'-ATCTCTCAATCCGCCTAGACGTC-3'
La mix della reazione è stata allestita utilizzando la GoTaq® DNA polimerasi e utilizzando le concentrazioni come di seguito indicato:
Figura 20. Mix di reazione e profilo termico di amplificazione
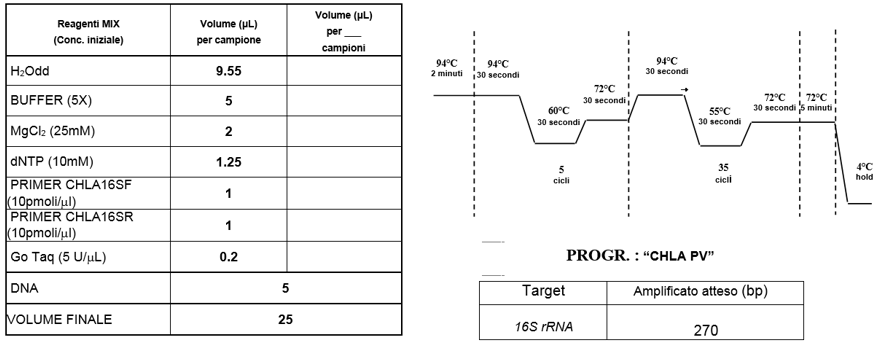
Figura 20. Mix di reazione e profilo termico di amplificazione
Figure 20. Reaction mix and thermal amplification profile
Gli amplificati vanno caricati su gel di agarosio al 2% e letti agli UV. Sarà considerato positivo il campione che avrà un amplificato di peso molecolare pari a 270 bp.
I campioni venuti positivi all'amplificazione con i primers specifici per Chlamydophila/Chlamydia possono essere sottoposti a digestione enzimatica con TRu1 I per l'identificazione di alcune specie. La reazione di digestione è da considerarsi correttamente eseguita quando i controllo positivi presentano un profilo di restrizione corrispondente a quello atteso come di seguito descritto:
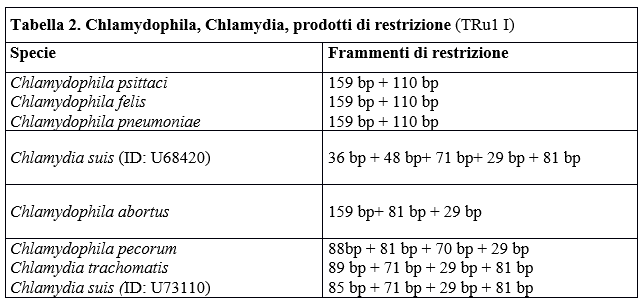
Table 2. Chlamydophila, Chlamydia, Restriction Products (TRu1I)
Per distinguere la Chlamydophila psittaci dalla felis e dalla pneumoniae si può usare l'enzima Hinf I. la reazione di digestione è da considerarsi correttamente eseguita quando i controlli positivi presentano un profilo di restrizione corrispondente a quello atteso:

Table 3. Restriction Products (Hinf I)
PCR per Toxoplasma gondii
L'esame, eseguito seguendo la letteratura (Magnino et al., 2000) procedura standard in uso presso il laboratorio di Diagnostica dell'IZSUM si esegue su un omogenato di organi (polmone e cervello).
Figure 21 a, b. Opening of skull and spinal canal
By IZSUM/2016 under a Creative Commons Attribution 4.0 International License (CC BY 4.0)
Figure 22. The lung (left) and brain (right) homogenate
By IZSUM/2016 under a Creative Commons Attribution 4.0 International License (CC BY 4.0)
Preparare una serie di provetta da 2 ml opportunamente identificate.
Porre in ciascuna provetta una biglia metallica e 1.2 ml di soluzione fisiologica.
Prelevare una porzione pari a 0.5 grammi dal polmone e dal cervello e porla nella provetta. Trasferire e provette negli alloggiamenti del Tissue lyser avendo cura di bilanciare i due alloggiamenti tra di loro.
Per omogeneizzare porzioni d'organo di queste dimensioni lo strumento va utilizzato ad una frequenza pari a 20 Hz per 10 minuti.
In ogni seduta di estrazione va estratto un campione negativo e uno positivo.
Per avere un controllo negativo di estrazione aggiungeremo 200 µl di Acqua bidistillata al posto della sospensione di organo; tale controllo ci permette di escludere contaminazioni in fase di estrazione. Come controllo positivo di estrazione invece verrà utilizzata una sospensione di organo già venuta positiva; tale controllo ci permette di garantire l'efficacia e l'efficienza dei reagenti e degli apparecchi utilizzati.
Per l'estrazione si utilizza il protocollo per fluidi biologici allegato al QIAmp DNA Mini Kit-QIAGEN:
- Porre 200 µl di Buffer AL in provette da 1.5 ml correttamente identificate.
- Prelevare 200 µl di sospensione dell'omogenato d'organo ed aggiungerlo alla provetta con il Buffer AL.
- Aggiungere ad ogni provetta 20 µl di proteinasi K.
- Vortexare le provette e incubare in un termomixer a 58°C per 10 minuti agitando a 1200 rpm.
- Spinnare velocemente le provette per rimuovere eventuali gocciolina dal tappo.
- Aggiungere 200 µl di Etanolo per arrestare l'azione della proteinasi K.
- Trasferire tutto il materiale dalle provette alle colonnine opportunamente identificate.
- Centrifugare le colonnine per 1 min. a 8000 rpm.
- Traferire le colonnine in nuovi tubi e aggiungere 500 µl di Buffer AW1 facendo attenzione a non toccare la colonnina con il puntale.
- Chiudere le colonnine e centrifugare per 1 min. a 8000 rpm.
- Al termine della centrifugata trasferire le colonnine su nuovi tubi.
- Aggiungere in ogni colonnina 500 µl di Buffer AW2 con le stesse modalità descritte prima.
- Centrifugare per 3 min. a 13500 rpm .
- Porre le colonnine su delle provette da 1.5 ml identificate ed eluire il DNA dalla colonnina aggiungendo 200 µl di Buffer AE. Lasciare il buffer a contatto per almeno 1 minuto. Centrifugare per 1 min. a 8000 rpm.
- Richiudere le provette contenenti il DNA che se non viene utilizzato subito va conservato a -20°C.
b) Amplificazione del DNA
La procedura in uso presso il laboratorio di Diagnostica dell'IZSUM è una PCR touch down. Si basa sull'amplificazione mediante PCR di una regione del gene codificante per il 18 S rRNA presente in Toxoplasma gondii.
Per ciascuna sessione di lavoro in cui vengono posti ad amplificare campioni in esame, vanno aggiunti un campione negativo di amplificazione (acqua bidistillata) e un campione positivo di amplificazione (DNA già estratto di un campione positivo già noto).
I primer utilizzati hanno le seguenti sequenze:
Primer COC 1: 5'-AAGTATAAGCTTTTATACGGC-3'
Primer COC 2: 5'-CACTGCCACGGTAGTCCAATAC-3'l
La mix della reazione è stata allestita utilizzando la GoTaq® DNA polimerasi e utilizzando le concentrazioni come di seguito indicato:
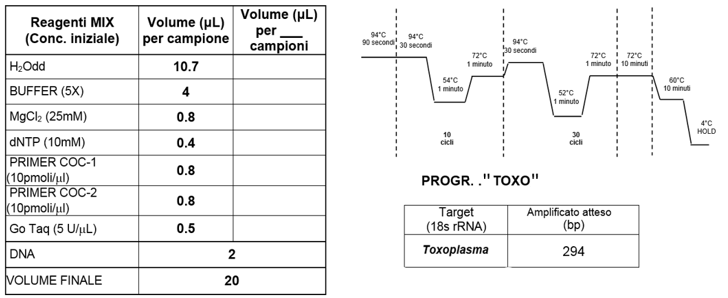
Figure 23 :. Reaction mix and thermal amplification profile
Gli amplificati vanno caricati su gel di agarosio al 2% e letti agli UV. Sarà considerato positivo il campione che avrà un amplificato di peso molecolare pari a 294 bp.
PCR per Criptosporidium spp. e Giardia duodenalis
Per la determinazione vengono seguite le indicazioni di letteratura (Gòmez-Couso et al., 2004).
a) Estrazione del campione
Viene impiegato un sistema di estrazione automatico, Nuclisens®, EasyMag Biomérieux, basato sulla tecnologia di estrazione con la silice (detta anche tecnologia di BOOM).
Durante l'incubazione dei campioni lisati con Lysis Buffer, contenente tiocianato di guanidina, l'acido nucleico veniva catturato da particelle di silice magnetica. Il dispositivo magnetico del Nuclisens® EasyMag attrae la silice, dando la possibilità al sistema di purificare gli acidi nucleici attraverso una fase di lavaggi ripetuti eseguiti mediante l'impiego di due tamponi differenti (NucliSENS® easyMAG®; http://www.biomerieux.ch/)
Al termine del ciclo di lavaggio segue una fase di riscaldamento atta a liberare gli acidi nucleici dalla silice ed una fase finale in cui le particelle di silice magnetica vengono separate dall'eluato attraverso il dispositivo magnetico.
b) Amplificazione del DNA
Criptosporidium spp. e Giardia duodenalis sono stati ricercati mediante l'utilizzo della multiplex-nested-PCR che ha come bersaglio il gene cowp (proteina di parete dell'oociste di Cryptosporidium) ed il gene tpi (trioso fosfato isomerasi) di Giardia duodenalis assemblage A e B.
La Multiplex-Nested-PCR utilizzata permette la ricerca simultanea di Cryptosporidium spp. e Giardia duodenalis assemblage A e B (Gómez-Couso et al., 2004). Nella prima PCR vengono amplificate una sequenza specifica di 430 bp del gene COWP e due sequenze specifiche del gene tpi di Giardia assemblage A e B di 576 bp e 210 bp, rispettivamente con i primers PCWOPF/PCOWPR, TPIAF/TPIAR e TPIBF/TPIBR (Tabella 4).
La reazione di PCR è stata effettuata in 10 l di volume di reazione, utilizzando 5 l di estratto di DNA in tampone per PCR 1 x, 2 mM di MgCl2, 250 M di ogni dNTP, 0,3 M di ogni primer e 0,05 U/ l di Taq DNA Polimerasi.
Le condizioni di amplificazione erano le seguenti: denaturazione iniziale a 94°C per 1 minuto, 25 cicli 94 C per 20 secondi, 52°C per 30 sec e 72 C per 1 minuto, seguiti da una estensione finale a 72 C per 5 minuti. Il prodotto della prima PCR è stato poi amplificato in tre reazioni separate di nested-PCR, di cui una di 341 bp specifica del gene COWP di Cryptosporidium, una di 452 bp specifica del gene tpi di G. duodenalis assemblage A e l'ultima di 141 bp specifica del gene tpi di G. duodenalis assemblage B (primers PCOWPIF/PCOWPIR, TPIAIF/TPIAIR e TPIBIF/TPIBIR, Tabella 2).
Le 3 reazioni di PCR sono state preparate in un volume finale di 20 l contenente 10 l di prodotto di PCR diluito (1/10 in acqua distillata sterile) e le seguenti concentrazioni:
tampone per PCR 1 x, 1,5 mM di MgCl2, 250 M di ogni dNTP, 1 M di ogni primer (PCOWPIF/PCOWPIR per gene COWP, TPIAIF/TPIAIR per Tpi assemblage A, TPIBIF/TPIBIR per Tpi assemblage B) e 0,1 U/ l di Taq DNA Polimerasi.
Le condizioni di amplificazione erano le seguenti per tutte e tre le reazioni: denaturazione iniziale a 94°C per 1 minuto, 25 cicli 94 C per 20 secondi, 52°C per 30 sec e 72 C per 1 minuto, seguiti da una estensione finale a 72 C per 5 minuti.
La Taq DNA polimerasi utilizzata per la PCR era la Go Taq Flexi DNA Polymerase con relativo tampone di reazione e MgCl2 (Promega). L'amplificazione in PCR è stata effettuata con un apparecchio termociclatore Mastercycler (Eppendorf).
Interpretazione dei risultati
Controlli positivi e negativi di reazione (DNA estratto da campione fecale positivo per Cryptosporidium spp. o da oocisti purificate di Cryptosporidium spp., DNA estratto da trofozoiti purificati per Giardia duodenalis assemblage A o B, acqua per PCR) sono stati inclusi per ogni analisi di PCR effettuata.
La tabella con i primer è la seguente:
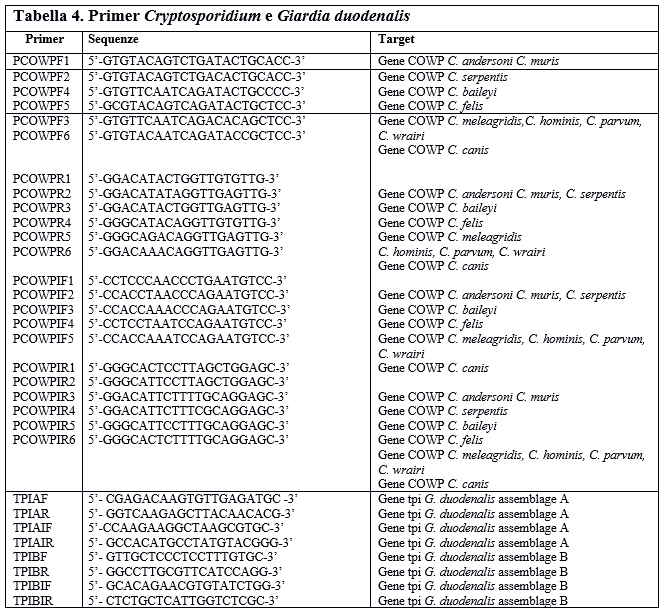
Table 4. Cryptosporidium and Giardia duodenalis Primers
|
ESAMI BATTERIOLOGICI
Gli esami batteriologici hanno riguardato:
- Ricerca Salmonella
- Ricerca Campylobacter
Ricerca Salmonella
La procedura tecnica per la ricerca di Salmonella dalle feci si riferisce alle indicazioni del Decreto del Ministero della Sanità del 10 marzo 1997 "Programma di controllo per le S. enteritidis e S. Typhimurium negli allevamenti di galline ovaiole destinate alla produzione di uova da consumo" ed al lavoro di Koneman (1995). Si basa su quattro fasi operative successive: pre-arricchimento, arricchimento selettivo, isolamento, identificazione biochimica e conferma sierologica, come specificato di seguito.
Figure 25 a, b: Intestinal examination - gastrointestinal block
By IZSUM/2016 under a Creative Commons Attribution 4.0 International License (CC BY 4.0)
1 Prearricchimento
Viene effettuato in terreno liquido non selettivo allo scopo di moltiplicare la flora batterica presente in origine favorendo lo sviluppo anche delle eventuali Salmonelle presenti in scarso numero o stressate.
2 Arricchimento selettivo
Viene effettuato in terreno liquido selettivo allo scopo di favorire la moltiplicazione di microrganismi del genere Salmonella inibendo quella di microrganismi diversi.
3 Isolamento
Viene effettuato utilizzando un terreno solido selettivo e differenziale per poter disporre di colonie isolate considerate tipiche o sospette.
4 Identificazione biochimica e conferma sierologica
L'identificazione di microrganismi appartenenti al genere Salmonella richiede l'allestimento delle seguenti prove biochimiche:
- Utilizzazione glucosio
- Utilizzazione lattosio
- Produzione di idrogeno solforato
- Produzione β-galattosidasi
- Crescita in presenza di solo citrato
- Decarbossilazione della lisina
- Idrolisi dell'urea
Viene effettuata mediante agglutinazione rapida su vetrino con sieri specifici anti-Salmonella del commercio.
Modalità di esecuzione della procedura
1 Prearricchimento non selettivo. Semina da campioni di feci e lettiera
- Contrassegnare un contenitore sterile con il n° di accettazione e la data e trasferire in esso 10 g ± 1 g di feci prelevate in pool.
- Aggiungere al materiale pesato BPW (Acqua peptonata tamponata) mantenendo il rapporto 1:10 p/v. Nel nostro caso ai campioni sono stati aggiunti 90 ml di acqua peptonata.
- Incubare la sospensione per non meno di 16 h e non più di 20 h a 37°C ± 1°C.
2 Arricchimento selettivo
- Contrassegnare una provetta contenente 10 ml di RV (Brodo Rappaport Vassiliadis) con il n° di accettazione, la data e l'indicazione del materiale esaminato.
- Dai brodi BPW seminare 0,1 ml in provette da 10 ml di RV.
- Incubare la provetta di RV a 42°C ± 1°C per 18-24 h.
3 Isolamento. Semina in piastra su terreni selettivi e differenziali
- Contrassegnare le piastre da utilizzare con il n° di accettazione, la data e l'indicazione del materiale esaminato.
- Prelevare una ansata di RV e seminare una piastra di XLD (Xylose Lysine Deoxycholate agar) e di Terreno cromogeno per Salmonella in maniera tale da ottenere colonie isolate.
- Incubare le piastre a 37°C ± 1°C per 18-24 h.
Lettura delle piastre
Al termine del periodo di incubazione verificare lo sviluppo di colonie tipiche.
Su XLD le colonie di Salmonella presentano centro nero e alone traslucido rossastro. Varianti negative a H2S cresciute su XLD sono rosa con centro rosa più scuro, mentre le Salmonelle lattosio positive su XLD sono gialle con o senza annerimento. Esaminare il terreno cromogenico per Salmonella ed identificare le colonie sospette, che si presentano di colore viola (rosso Magenta).la Salmonella sviluppa colonie rosse con viraggio del terreno circostante al rosso vivo, a margini interi, lucide, dal diametro di 2-3 mm.
4 Identificazione biochimica e conferma sierologica
Procedura per l'Identificazione delle colonie sospette.
Contrassegnare tre provette di TSA (Typtone soya agar) da utilizzare con il n° di accettazione, la data e l'indicazione del materiale esaminato e seminare complessivamente tre colonie ben isolate, che presentano caratteristiche morfologiche tipiche. Qualora non fossero disponibili tre colonie ben isolate, trapiantare tutte quelle presenti nelle piastre.
Incubare a 37°C ± 1°C per 18-24 h.
L'identificazione biochimica di genere della Salmonella può essere eseguita tramite un sistema in macrometodo o utilizzando i sistemi di identificazione in micrometodo del commercio seguendo le istruzioni del produttore per quanto riguarda le modalità di esecuzione delle prove. Il laboratorio utilizza il Kit RapiD 20 ETM della Ditta Biomerieux che permette di avere i risultati in 4 ore.
Il sistema consiste in gallerie preconfezionate costituite da 20 microprovette che contengono dei substrati disidratati. La microprovette vengono inoculate con una sospensione batterica pari a 0.5 MC Farland diluita in soluzione fisiologica. Durante il periodo di incubazione le reazioni prodotte producono un viraggio di colore. La lettura di queste reazioni deve essere fatta attraverso i criteri interpretativi riportati nelle istruzioni del kit e si traduce in un profilo numerico.
Il codice numerico viene identificato dal software della Ditta che è disponibile al sito https://apiweb.biomerieux.com
Qualora l'identificazione fosse negativa per Salmonella sul primo TSA, ripetere l'identificazione per le restanti piastre di TSA.
Tipizzazione Sierologica
I ceppi identificati come Salmonella spp. vengono sottoposti alla tipizzazione sierologica mediante agglutinazione rapida su vetrino presso il laboratorio di Riferimento Regionale per gli Enteropatogeni di Perugia.
|
Ricerca Campylobacter
La procedura in uso presso il laboratorio di Diagnostica dell'IZSUM per l'identificazione dei Campylobacter termotolleranti si riferisce ai lavori di (Quinn et al., 1999; Pezzotti et al., 2003). A partire da materiale fecale segue tre fasi operative:
1. arricchimento selettivo
2. isolamento
3. identificazione.
1 Arricchimento selettivo
Contrassegnare un sacchetto per agitatore peristaltico (Stomacher) con il numero di accettazione e la data e trasferire in esso 5 g di feci ± 5% e aggiungervi 45 ml ± 5% di Preston brodo. Agitare manualmente la sospensione ottenuta e incubare a 41,5°C ± 1°C per 44 h ± 4h in microaerofilia.
2 Isolamento
Prelevare un'ansata di brodocoltura ed inoculare una piastra di terreno di Karmali effettuando una semina per diluizione, in modo da ottenere colonie isolate.
Dopo la semina capovolgere le piastre esaminate ed incubarle a 41,5°C ± 1°C per 44 h ± 4h.
Al termine del periodo di incubazione verificare lo sviluppo di colonie tipiche:
Su karmali agar i Campylobacter termotolleranti sviluppano colonie biancastre, di aspetto mucoide, con schiarimento del terreno circostante.
Prelevare mediante ago sterile, tre colonie isolate considerate tipiche o sospette.
Seminare ogni colonia selezionata su una superficie di una piastra di Agar Sangue ed incubare le piastre capovolte in termostato a 41,5 °C ± 1°C in condizioni di microaerofilia.
3 Identificazione
Le colture pure in AS vanno identificate valutandone la morfologia e le reazioni biochimiche.
Colorazione di Gram:
I germi isolati vanno colorati mediante colorazione di Gram; al microscopio ottico con l'obiettivo ad immersione (ingrandimento 100X ) i Campylobacter si presentano come bastoncelli gram negativi, disposti a virgola, a spirale o ad "ali di gabbiano".
Test della catalasi:
Utilizzando un vetrino porta-oggetto mettere a contatto un'ansata di patina batterica con una goccia di perossido d'idrogeno. Il test si considera positivo se si sviluppano bollicine di gas. I microrganismi del genere Campylobacter sono catalasi positivi.
Test dell'ossidasi:
il test si effettua a partire da un'ansata di patina batterica, utilizzando un kit in commercio.
Il laboratorio utilizza il BBL Dryslide TM della Becton Dickinson.
I microrganismi del genere Campylobacter sono ossidasi positivi.
4 Interpretazione dei risultati
Vengono considerati positivi campioni che abbiano dato luogo allo sviluppo di colonie che presentino caratteristiche morfologiche tipiche del genere Campylobacter, ossidasi positive e catalasi positive.
Le colonie isolate vengono poi ulteriormente analizzate per procedere alla caratterizzazione di specie mediante PCR.
|
Multiplex PCR
La procedura in uso presso il laboratorio di Diagnostica dell'IZSUM è una multiplex PCR che si basa sulla contemporanea amplificazione dei geni di alcune specie di Campylobacter spp (C. Coli, C. Lari, C. Upsaliensis, C. Fetus subsp. fetus e C. Jejuni) in un'unica reazione di PCR (multiplex) mediante l'impiego di primers specifici.
Estrazione del DNA per bollitura
Prelevare una o più colonie batteriche con un'ansa monouso sterile e porla in provetta da 1,5 ml sterile contenente 200 ml di Soluzione fisiologica.
Bollire per 10-15 minuti
Centrifugare brevemente a circa 8000 rpm per 1 minuto
Utilizzare il surnatante.
Per ciascuna sessione di lavoro in cui vengono posti ad amplificare campioni ignoti, vanno aggiunti un campione negativo di amplificazione (acqua bidistillata) e un campione positivo di amplificazione (DNA già estratto di un campione positivo già noto di uno o più Campylobacter).
I primers utilizzati hanno le seguenti sequenze:
Primer:
- CJ Forward: 5' ACTTCTTTATTGCTTGCTGC 3'
- CJ Reverse: 5' GCCACAACAAGTAAAGAAGC 3'
- CC Forward: 5' GTAAAACCAAAGCTTATCGTG 3'
- CC Reverse: 5' TCCAGCAATGTGTGCAATG 3'
- CL Forward: 5' TAGAGAGATAGCAAAAGAGA 3'
- CL Reverse: 5' TACACATAATAATCCCACCC 3'
- CU Forward: 5' AATTGAAACTCTTGCTATCC 3'
- CU Reverse: 5' TCATACATTTTACCCGAGCT 3'
- CF Forward: 5' GCAAATATAAATGTAAGCGGAGAG 3'
- CF Reverse: 5' TGCAGCGGCCCCACCTAT 3'
- 23S Forward: 5' TATACCGGTAAGGAGTGCTGGAG 3'
- 23S Reverse: 5' ATCAATTAACCTTCGAGCACCG 3'
La mix della reazione è stata allestita utilizzando la GoTaq® DNA polimerasi e utilizzando le concentrazioni come di seguito indicato:
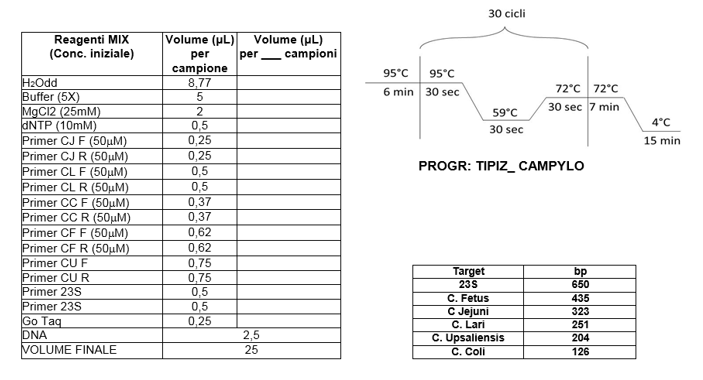
Fig. 26: Reaction parameters with GoTaq DNA Polymerase
La lettura dei risultati viene effettuata verificando la presenza/assenza di amplificati, le cui dimensioni vengono confrontate con quelle del/i controllo/i di amplificazione e con quelle del marcatore di peso molecolare.
|
RISULTATI
In relazione alle attività di monitoraggio condotte, in vista di azioni finalizzate al contenimento della popolazione di piccioni urbani, sono stati effettuati da Marzo a Luglio del 2016, prelievi di animali al fine di valutarne le condizioni sanitarie ed il rischio zoonosico che la loro presenza comporta.
Tali dati sono stati comparati con quelli forniti dall' Osservatorio Epidemiologico Veterinario dell'IZS UM già oggetto di pubblicazioni nel corso del tempo (Montefameglio et al., 2003; Grelloni et al., 2007; Maresca et al., 2011).
Di seguito sono riportate le tabelle relative ai lavori già svolti e ai risultati di queste ultime indagini (2016) su Salmonella spp, Campylobacter termofili, Clamydia psittaci, Toxoplasma gondii e endoparassiti quali: Trichomonas spp, Capillaria spp, ascaridi, tenie e coccidi.
Salmonella
L'isolamento di Salmonella mostra un andamento altalenante. Nel 2003 non è stata rinvenuta esaminando 150 campioni. Nel 2007 invece, su 142 campioni esaminati, è stata isolata da 4 campioni (prev. 3%) . Dai 75 campioni esaminati nel 2011 la Salmonella è stata isolata solo da 1 animale (prev.1,33) . Nel nostro lavoro condotto nel 2016 non è stata riscontrata la presenza di Salmonella degli animali esaminati.
Campylobacter termotolleranti
I nostri rilievi, condotti sugli animali catturati nel 2016, confermano l'assenza di Campylobacter coli già, peraltro citata in tutti gli altri lavori con i quali ci siamo confrontati.
Il C. jejuni nel 2003 è stato isolato in 4 animali (prev.2,6%); nel 2007 la prevalenza è salita al 16% avendo isolato C. jejuni in 23 animali.
Nel 2011 gli animali risultati positivi sono stati 5 riducendo la prevalenza al 6,7%. Gli isolamenti effettuati sui 41 animali, oggetto del presente studio, non hanno dato alcun esito positivo (prev. 0%). Di seguito riportiamo le relative tabelle.

Table 5. Detection of Salmonella spp and thermophilic
Campylobacter - by Montefameglio et al., 2003, modified (number and prevalence%)

Table 6. Results of the isolations of
Salmonella spp and thermophilic
Campylobacter - by Grelloni et al., 2007, modified (number and% prevalence)
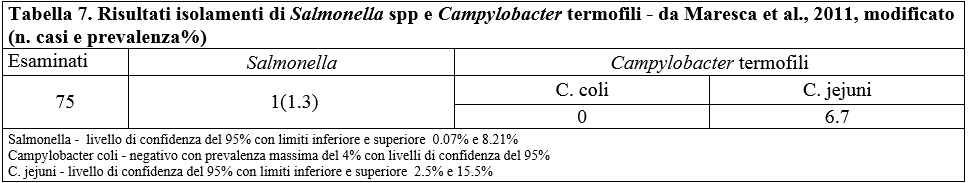
Table 7. Isolation results of Salmonella spp and thermophilic
Campylobacter - by Maresca et al., 2011, modified (number of cases and% prevalence)

Table 8. Isolation results of Salmonella spp and thermophilic
Campylobacter - 2016 (number of cases and prevalence%)
Chlamydophila
Le ricerche condotte per Clamydophila psittaci fino al 2011 sono state di tipo indiretto.
La prevalenza di animali positivi per la presenza di anticorpi nei confronti di Clamydophila ha seguito lo stesso andamento riscontrato per le infezioni da Salmonella e C. jejuni.
La prevalenza di positivi ha oscillato dal 35% nel 2003 al 24% nel 2007 ed è scesa al 24% nel 2011. Grazie alla riduzione dei costi delle analisi molecolari e per valutare l'effettivo potenziale zoonosico degli animali ritrovati positivi, nel corso di questo lavoro si è deciso di effettuare una ricerca diretta di tale patogeno tramite PCR trovando un animale positivo sui 41 esaminati (prev. 2.4%).
Toxoplasma
I lavori precedenti per l'infezione da Toxoplasma gondii hanno utilizzato metodiche di tipo indiretto (ELISA) mostrando valori di prevalenza oscillanti dal 18% all'83%. La ricerca diretta di tale protozoo, condotta durante la nostra indagine non ha rilevato animali positivi alla PCR. Di seguito riportiamo le relative tabelle.

Table 9. Serological Survey Results - by Montefameglio et al., 2003, modified (number of positive cases and% prevalence)

Table 10. Serological Survey Results - by Grelloni et al., 2007, modified (number of positive cases and% prevalence)

Table 11. Serological Survey Results - by Maresca et al., 2011, modified (number of positive cases and % prevalence)
Endoparassiti
Il confronto del carico parassitario riscontrato in tutti i lavori esaminati identifica il 2011come con il valore più elevato. I valori riscontrati in questo ultimo campionamento, invece, sembrano essere i minori (ad eccezione degli ascaridi) rispetto ai precedenti, come si evince dalle tabelle.
Tali risultati possono essere attribuiti sia alla scarsa consistenza campionaria negli anni 2011 e 2016, che agli effetti delle campagne di "sfoltimento" delle colonie di piccione urbano, effettuate dalla amministrazione del Comune di Perugia. Di seguito riportiamo le relative tabelle.
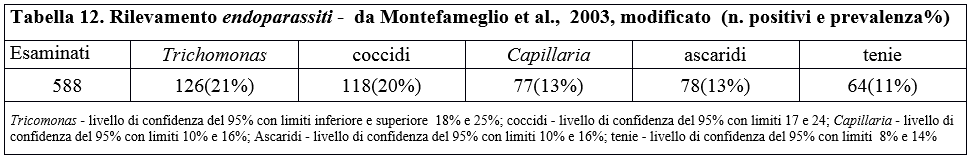
Table 12. Endoparasite detection - by Montefameglio et al., 2003, modified (number of positive cases and % prevalence)
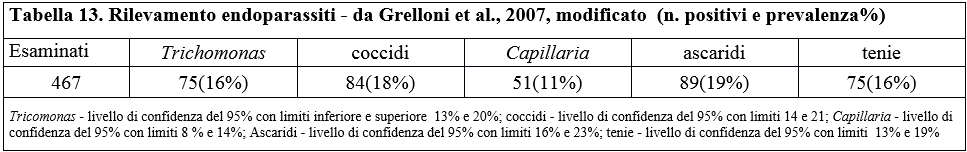
Table 13. Endoparasite detection - by Grelloni et al., 2007, modified (number of positive cases and % prevalence)
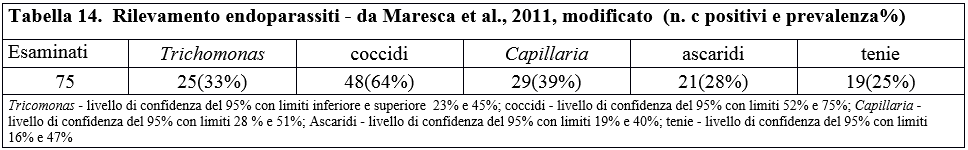
Table 14. Endoparasite detection - by Maresca et al., 2011, modified (number of positive cases and % prevalence)
![Tabella 15. Rilevamento endoparassiti (n. positivi e prevalenza%) [per cortesia dell'Istituto Zooprofilattico Sperimentale UM, 2016]](immagini/tamTAB015.png)
Table 15. Endoparasite detection (number of positive cases and % prevalence) [Courtesy of Zooprofilattico Sperimentale Institute of Umbria and Marche, 2016]
Nuove determinazioni effettuate nell'indagine del 2016
Nel corso di questo lavoro si è indagato anche sulla presenza di patogeni che non sono stati ricercati nei precedenti anni. Non si sono riscontrate positività per Cryptosporidium / giardia sui pool di feci animali, ne' per Cryptococcus e miceti sui polmoni singoli. Anche la ricerca di Coxiella burnetii su pool di organi ha dato esito negativo. Di seguito riportiamo le relative tabelle.
![Tabella 16. Risultati PCR da pool di feci [IZSUM, 2016]](immagini/tamTAB016.png)
Table 16. PCR results from faeces pool [IZSUM, 2016]
![Tabella 17. Risultati colturali da pool di organi [IZSUM, 2016]](immagini/tamTAB017.png)
Table 17. Growt of
Toxoplasma gondii from organ pool [IZSUM, 2016]
![Tabella 18. Risultati colturali da pool di organi [IZSUM, 2016]](immagini/tamTAB018.png)
Table 18. Growth of
Candida albicans and
Cryptococcus spp from organ pool [IZSUM, 2016]
DISCUSSIONE E CONCLUSIONI
L'andamento della Salmonella mostra una situazione altalenante. Nel 2003 non è stata rinvenuta, esaminando una quantità rilevante di campioni (150). La positività di 3 animali rilevata nel 2007 non viene confermata nel campionamento del 2016, mostrando come le condizioni siano via via migliorate nel tempo. Per quanto riguarda il Campylobacter coli non si rilevano positività nel periodo. Di maggiore interesse risulta l'andamento del Campylobacter jejuni che mostra un'analogia con quanto abbiamo osservato per la Salmonella.
La stessa osservazione vale per Clamydia psittaci che, dai dati sierologici ricavati, mostra una situazione analoga al Campylobacter jejuni. Il potenziale zoonosico di tale patogeno viene confermato dalla positività in PCR osservata nel lavoro del 2016.
In sintesi, pur essendo la presenza di Clamydia e Campylobacter jejuni significativamente maggiore rispetto a Salmonella e Campylobacter coli, il loro comune declino dal 2007 ad oggi, sembra indicare la non urgenza di mettere in campo azioni preventive per evitare possibili zoonosi da esse derivanti.
La Clamydia psittaci, riscontrata nel corso del nostro studio, è responsabile della psittacosi, la cui trasmissione avviene prevalentemente per inalazione di escrementi essiccati dei volatili (nell'uomo può causare una polmonite interstiziale con un andamento clinico grave). Quindi, sia pure in presenza di un rischio limitato, tale positività deve comunque destare interesse in ambito sanitario.
Figura 27: Andamento rilevamenti su Salmonella, Clamydia, Campylobacter coli,
Campylobacter jejuni dal 2003 al 2016
Situazione diversa è invece quella definita dai parassiti che rappresentano invece un tratto caratteristico della popolazione di piccioni studiata nel presente lavoro.
Figura 28: Andamento infestazioni da Tricomonas, coccidi,
Capillaria, ascaridi, tenie, dal 2003 al 2016
Dall'andamento del grafico possiamo infatti notare come sin dall'inizio la presenza dei parassiti, soprattutto Trichomonas e coccidi, sia maggiore rispetto alle Tenie e che il loro andamento veda un forte incremento intorno al 2011 (coccidi), con un rapido declino fino ai giorni nostri, mentre la presenza di Ascaridi si mantiene costante.
Nonostante la diffusa positività dei test microbiologici e parassitologici effettuati nel corso del tempo da vari soggetti, "pochissimi sono i lavori tesi a chiarire il ruolo di questa specie nell'epidemiologia delle infezioni umane o di altre specie animali" (Mancini 2002). Questa affermazione spinge a intensificare le valutazioni derivanti da dati rilevati direttamente o desunti da altri lavori. Nel nostro caso le indagini effettuate dal 2003 al 2016, che sono state citate, mostrano come una corretta gestione della popolazione di Columba livia in Umbria, abbia ridotto il rischio di trasmissione di malattie a carattere zoonotico.
Le indagini relative ai parassiti sono state effettuate per valutare lo stato sanitario del piccione urbano; la riduzione della carica parassitaria testimonia una corretta gestione, soprattutto dal punto di vista numerico, di tale popolazione.
Per quanto riguarda i parassiti, Giardia duodenalis, e Cryptosporidium spp, specie pericolose anche per l'Uomo, non sono risultate presenti, come pure i miceti (Cryptococcus spp e Candida albicans).
La possibile presenza di altri parassiti, pure molto importanti quali zecche (trasmissione all'Uomo di Spirochete e Rickettsie) e pidocchi, non sono stati riscontrati nei 41 animali esaminati nel 2016; è importante comunque continuare una sorveglianza attiva/passiva relativa agli ectoparassiti citati.
Come visto, i rischi sanitari batterici e parassitari, sono derivanti prevalentemente dalla popolazione di piccioni e dalla contaminazione fecale dell'ambiente, con materiali che si disperdono per via anemofila in aree anche molto estese.
La nostra esperienza conferma l'efficacia del contenimento numerico delle popolazioni di volatili urbani, tale attività può essere svolta tramite cattura, come nel nostro caso, o tramite dissuasori e sistemi che impediscono o scoraggiano l'insediamento di massa in aree circoscritte.
Possono essere utilizzati vari deterrenti.
Animali, rappresentati da rapaci.

Figura 30. I falchi (in questa foto un ibrido), possono essere utilizzzati come deterrente per i piccioni.
Tuttavia perché siano efficaci è necessaria una presenza rinnovata frequentemente nella zona da proteggere
Figure 30. Hawks (in this photo a hybrid) may be used as a deterrent for the pigeons.
However, in order to be effective, a frequently renewed presence in the area to be protected is required
Foto R. Ciappelloni (2016). Licenza Creative Commons. Attribution-NonCommercial 4.0 International [CCBYNC4.0]
Deterrenti visuali come oggetti di diversa foggia e dimensione (visual scare, balloon repels), effetti luminosi (laser bird repeller), sonori (sonic/ultrasonic pest bird repeller), device elettrici (fanno uso di correnti a bassa intensità: <0.5 joule), sostanze chimiche repellenti o collanti.
Altri sistemi prevedono la sterilizzazione mirata di uccelli praticata con sistemi chirurgici (vasectomia bilaterale nel maschio) o chimici come busulfan®, ornitrol® o nicarbazina (quest'ultima da alcuni criticata per la sua presunta pericolosità).
Tali interventi si collocano fra le attività che necessitano di una concertazione fra servizi sanitari ed autorità regionali e municipali.
La scelta della cattura come metodo di contenimento è l'unica che permette un monitoraggio attivo degli agenti zoonotici presenti sul territorio. Un ulteriore accorgimento per contenere il rischio zoonosico è quella di rimuovere sistematicamente la fonte di infezione rappresentata da accumuli di guano che si formano sui tetti e nei compluvi.
La rimozione delle feci evita che, tramite l'uso di aspiratori e ventilatori, le polveri di deiezioni secche possano facilmente entrare negli appartamenti, ristoranti, uffici, ospedali, scuole infettando gli ambienti.
Se si riuscisse ad introdurre e sostenere una corretta gestione delle popolazioni di questo sinantropo, sarebbe forse possibile organizzare una utile convivenza fra Columba livia e altre specie (umana, animali di allevamento e da compagnia), anche considerando che il piccione è ormai entrato stabilmente a far parte dell'arredo urbano.
In considerazione di ciò, per la sua diffusa presenza questi dovrebbe essere considerato non solo come possibile vettore di zoonosi, ma anche come importante bioindicatore di inquinamento ambientale.
Bibliografia
Alexander D. J., Wilson G. W., Russell P. H. , Lister S. A., Parsons G. (1985) Newcastle disease outbreaks in fowl in Great Britain during 1984. Veterinary Record, 117: 429-434.
Amadori M. L., Gramegna E., Raffaelli G., Schillaci F., Mangani F., Bachiocca I. (2003). La facciata della chiesa di santo spiritoin urbino: dalle indagini scientifiche al restauro. Quaderni di Scienza della Conservazione. Bologna.
Ambrosi M. (1995). Parassitologia Zootecnica. Edizioni Agricole. Bologna.
Andreoni S., Farina C., Lombardi G. (2003). Atlante di Micologia Medica. Grafik@rt srl, Paderno Dugnano.
Berri M., Laroucau K., Rodolakis A. (2000) "The detection of Coxiella burnetii from ovine genital swabs,milk and fecal samples by the use of a single touchdown polymerase chain reaction " Veterinary Microbiology. 72: 285-293.
Boch J., Superer R. (1980). Parassitologia Clinica Veterinaria, Essegivi, Piacenza
Boom R., Sol J., Salimans M. M., Jansen C. L., Wertheim-Van Dillen P. M., Van der Noordaa J. (1990). Rapid and simple method for purification of nucleic acids. Journal of Clinic Microbiology. 28(3): 495-503.
Gargiulo A., Pasqualina Russo T., Schettini R., Mallardo K., Calabria M., Menna L. F., Raia P., Pagnini U., Caputo V., Fioretti A., Dipineto L. (2014). Occurrence of Enteropathogenic Bacteria in Urban Pigeons (Columba livia) in Italy. Vector-borne and zoonotic diseases. Volume 14, Number 4.
Gómez-Couso H., Freire-Santos F., Amar C.F.L., Grant K.A., Williamson K., Ares-Mazás M.E., McLauchlin J. (2004). Detection of Cryptosporidium and Giardia in molluscan shellfish by multiplexed nested-PCR. Int. Journal of Food Microbiology. 91,279-288.
Grelloni V, Costarelli S, Crotti S, Calzoni P, D'Angelo G, Mariotti C, Scoccia E, Venditti G, Maresca C. (2007). Sorveglianza della popolazione dei piccioni (Columba livia) nella città di Perugia. Congresso: IX Congresso Nazionale SIDILV Roma 14-16 novembre.
Haag-Wackernagel D., Moch H. J. (2004). Health hazards posed by feral pigeons. Infect. 2004 May;48(4):307-13.
Haag-Wackernagel, D. (2011). Vom Liebling der Go¨ tter zur Eroberung der Sta¨dte Die Taube*eine Erfolgsgeschichte. Biologie in unserer Zeit [The pigeon - a success story], 41, 44_52.
Horvatek Tomi? D., Laroucau K., Prukner-Radov?i? E.(2013). Detection of Chlamydia psittaci genotypes in fecal samples of homing pigeons in Croatia. Veterinarski Arhiv 83 (2), 201-209.
Jerolmack C. (2008). How Pigeons Became Rats: The Cultural-Spatial Logic of Problem Animals. Social Problems. Vol. 55, No. 1 (February), pp. 72-94.
Koneman E.W. (1995). Testo atlante di Microbiologia diagnostica. II edizione italiana, Antonio Delfino Editore, Roma.
Magnino S., Vicari N., Boldini M., Rosignoli C., Nigrelli A., Andreoli G., Pajoro M., Fabbri M. (2009) "Rilevamento di Coxiella burnetii nel latte di massa di alcune aziende bovine lombarde" Large Animal Review 15: 3-6.
Magnino S., Vigo P.G., Bandi C., Rosignoli C., Boldini M., Vezzoli F., Alborali L., Cammi G., Foni E., Colombo N., Colombo M., Bergami C., Mellini A., Fabbi M., Genchi C. (2000). La Selezione Veterinaria, Suppl, S15-S23.
Mancini S., 2002. La gestione delle popolazioni di colombi di città: aspetti giuridici e principali metodi di contenimento. Sanità Pubblica Veterinaria, n. 11/12, Febbraio, http://indice.spvet.it#74.
Maresca C., Boto S., Calzoni P., Caporali A., Checcarelli S., D'Avino N., Scoccia E., Tentillini M., Grelloni V., (2011). Popolazione dei piccioni (Columba livia) a Todi (PG): lo stato sanitario - Population of pigeons (Columba livia) in Todi city (Italy): health status. Sanità Pubblica Veterinaria, n. 64, Febbraio, http://indice.spvet.it#512.
Montefameglio M., Maresca C., Magistrali C., Costarelli S., Moscati L., Grelloni V., 2003. Popolazioni di piccione urbano: interventi finalizzati al risanamento del centro storico di Perugia e controlli sanitari. Webzine Sanità Pubblica Veterinaria, n. 22, Dicembre, http://indice.spvet.it#182.
Ossewaarde, J. M. and Meijer, A. (1999). Molecular evidence for the existence of additional members of the order Chlamydiales. Micro biology 145: 411-417.
Pezzotti G., Serafin A., Luzzi I. , Mioni R., Milan M., Perin R. (2003). Occurrence and antimicrobial resistance of Campylobacter jejuni and Campylobacter coli in animals and meat in north-eastern Italy. International Journal of Food Microbiology. 82, 281-287.
Polonelli L., Ajello L., Morace G. (1994). Micologia Medica. Società Editrice Esculapio, Milano.
Quinn P. J., Carter M. E., Markey B., Carter G. R. (1999). Clinical Veterinary Microbiology. Mosby publish ring London.
Taccini E., Rossi G., Gili C., (2006). Tecnica autoptica e diagnostica cadaverica. Manuale pratico di Veterinaria. Poletto Editore. Milano.
Vicari, N., Santoni, R., Vigo, P.G., Magnino, S. (2004). A PCR-RFLP assay targeting the 16S ribosomal gene for the diagnosis of animal chlamydioses. Proceedings, 5th Meeting of the European Society for Chlamydia Research (Ed.: Judith Deak), Budapest, Hungary, 1-4 September 2004, p. 297.
Vidotto V. "Manuale di Micologia Medica", 2002, Masson S.p.A., Milano.
Vogelstein B., Gillespie D. (1979). Preparative and analytical purification of DNA from agarose. PNAS USA (1979) 76(2): 615-619.

_________________________________________
Si ringraziano:
Raoul Ciappelloni, Carmen Maresca, Ylenia Abbate, Andrea Valiani, Silvia Crotti, Luigi Spaccini.

Tammaro et al., 2017. Aspetti sanitari della popolazione urbana di
Columba livia in Umbria. (SPVet.it 100/2017)





































![Tabella 15. Rilevamento endoparassiti (n. positivi e prevalenza%) [per cortesia dell'Istituto Zooprofilattico Sperimentale UM, 2016]](immagini/tamTAB015.png)
![Tabella 16. Risultati PCR da pool di feci [IZSUM, 2016]](immagini/tamTAB016.png)
![Tabella 17. Risultati colturali da pool di organi [IZSUM, 2016]](immagini/tamTAB017.png)
![Tabella 18. Risultati colturali da pool di organi [IZSUM, 2016]](immagini/tamTAB018.png)



